NP精读丨石斛属超级泛基因组研究
兰花是被子植物中最大且最为多样化的科之一,石斛属(Dendrobium Sw.)是兰科三大属之一,包含约1800个物种。石斛属物种表现出高度多样化和复杂的形态,由于其观赏和药用价值,石斛在全球范围内引起了公众和研究人员的广泛关注和兴趣,成为许多国家和地区的重要经济植物。
最近,来自新加坡和中国的研究团队合作在著名期刊Nature Plants上面发表了名为《Pangeneric genome analyses reveal the evolution and diversity of the orchid genus Dendrobium》的研究论文,文章通过测序和分析17个染色体级别的高质量基因组构建了石斛属超级泛基因组,通过解析一个石斛属杂交种单倍型基因组,揭示了杂合基因组中的单倍型变异和等位基因不平衡。在石斛属泛尺度分析中的发现极大地揭示兰科植物起源、进化和多样化的奥秘,同时也显著促进了石斛育种中遗传多样性的有效利用。

一、石斛杂交种单倍型基因组
石斛属杂交二倍体“Chao Praya Smile”(2n = 2x = 38)的谱系来源于石斛属中的8个物种。作者通过PacBio HiFi测序和Hi-C测序构建了这个杂交种单倍型基因组,通过共线性分析识别两个单倍型上存在大量的SNP、Indel和SV变异。其中38%的SVs与3,486个基因的编码区及侧翼区域(2 kb)重叠,表明SVs在基因调控中可能具有潜在作用。例如,两个石斛单倍型中的WSD11同源基因在内含子区域包含两个PAVs,并在萼片、花瓣和唇瓣中表现出不同的表达水平。
随后作者发现1,476个基因在所有组织中始终偏向一个等位基因表达(conASE),126个基因在不同等位基因之间切换高表达(inASE),以及6,704个AEE基因。而与AEE基因相比,ASE基因中SVs的发生频率更高,这突显了杂交石斛兰从亲本物种继承的多态性所产生的高度发育可塑性。
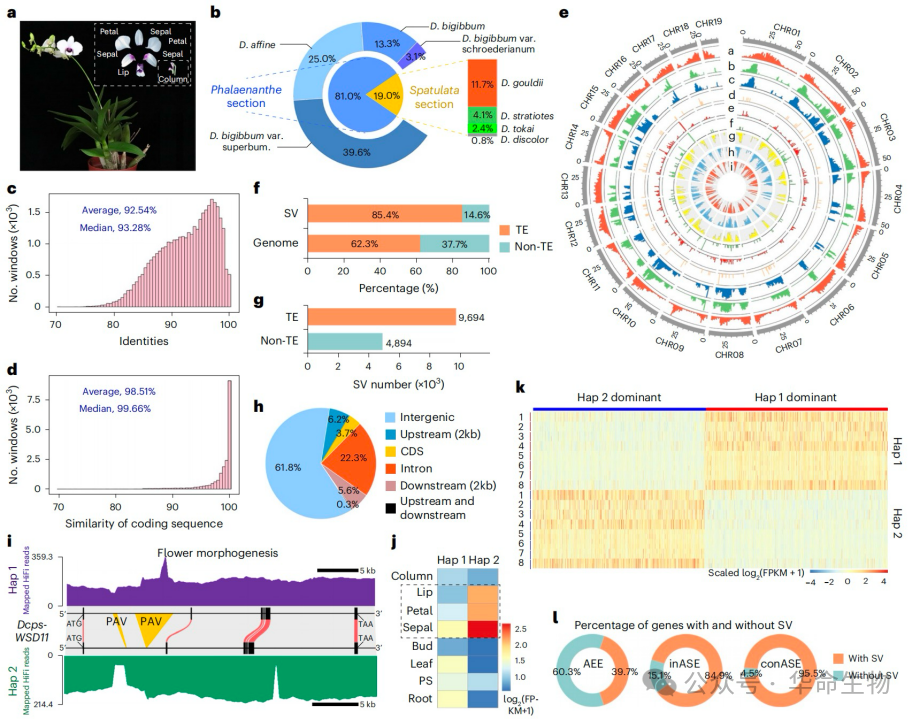
图1:石斛杂交种的基因组分析
二、13个石斛属物种基因组
为了全面探索石斛属兰花的基因组多样性,作者从12个主要石斛属组中选择了16个物种,其中3个已有发表的染色体级别参考基因组,作者对剩余的13个物种以及杂交种“Chao Praya Smile”分别进行了PacBio HiFi、Hi-C和Illumina测序。随后,对每个样本进行了denovo组装,得到的contig N50为5.71至25.39 Mb(平均14.08 Mb),scaffold N50为39.08至100.29 Mb(平均60.31 Mb)。BUSCO评估、LAIs和Merqury评估显示14个基因组组装均具有高准确性、完整性和连续性。
注释分析显示组装的基因组中平均识别出25,122个蛋白质编码基因,范围从20,004到31,440个。TEs在这些基因组中平均占58.66%,范围从52.99%到65.88%。
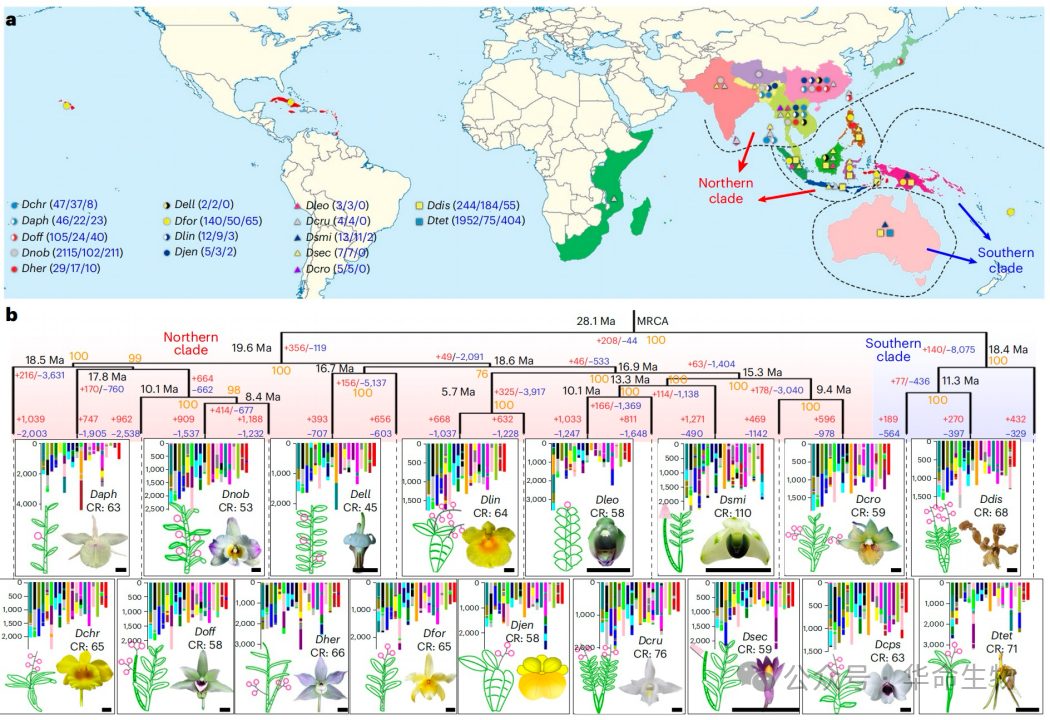
图2:17个石斛属物种基因组情况
三、石斛属演化历史分析
除了所有现存兰花物种共有的一次全基因组复制(WGD)事件外,在石斛属中未检测到额外的WGD。作者构建了一个高可信度的系统发育树,并使用来自27个不同植物物种的单拷贝直系同源基因家族计算了分歧时间。收缩和扩张基因家族分析显示扩张的基因家族可能增强了石斛属对广泛的地理和气候变化的适应能力。
尽管石斛属被认为包含两个主要分支(北方分支和南方分支),但其最早祖先的确切年龄仍存在争议。根据文章系统发育分析表明,它们的分歧可以追溯到2810万年前,此外,文章还澄清了多个之前分类可能错误的物种情况。分子系统发育与基于形态特征和地理位置的传统分类之间的这种差异,说明由于石斛属惊人的物种多样性,建立连贯的分类系统非常复杂。
四、超级泛基因组和CNSs分析
作者利用蛋白质编码基因进行了石斛属超级泛基因组分析。在所有泛基因簇中,23.99% 为核心基因簇,15.00%是软核心基因簇,49.28%为可缺失基因簇,11.73%为特有基因簇。核心和软核心基因的Ka/Ks比值低于可缺失基因和特有基因,且Pfam注释率更高,这表明核心和软核心基因在进化上更为保守。另外可缺失基因和特有基因的转座元件(TE)覆盖度高于核心和软核心基因,这表明TE在产生基因变异性方面发挥着更为活跃的作用。
此外,作者发现,4024 个核心基因含有石斛属特异性的保守非编码序列(CNSs),并且这些基因在与脂肪酸代谢和碳代谢相关的通路中显著富集,而这两种代谢对于能量产生和植物生长均至关重要。
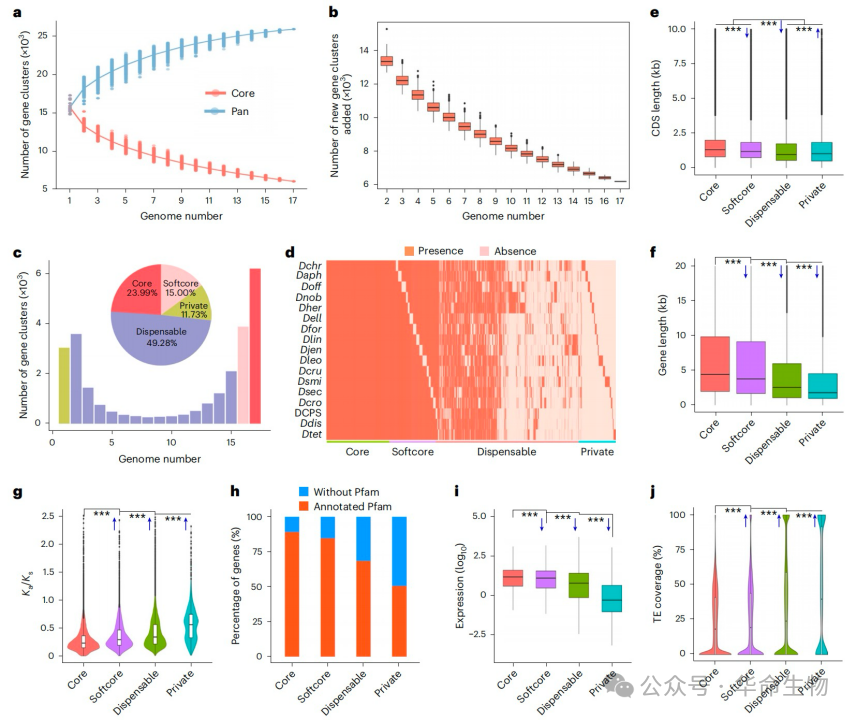
图3:石斛属super-pangenome构建
五、石斛属基因组倒位研究
作者通过对滇桂石斛(D. lindleyi)与其他15个来自不同谱系的基因组进行两两比对,在属的尺度上共鉴定出49,701个非冗余倒位,平均每物种有16Mb的基因组序列受到影响。倒位的长度在1kb到62kb之间,平均长度为2.3kb。
作者发现,倒位区域的TE含量显著低于周边区域。对倒位内基因的Ka/Ks以及Pfam注释率的分析表明,与非倒位区域相比,倒位区域具有更为保守和古老的起源。共享倒位和特异性倒位主要与核心基因和软核心基因相关,GO和KEGG富集分析显示,共享倒位或特异性倒位中的基因在基本生物学过程中富集,而非倒位区域的基因主要与应激防御相关的次生代谢产物、植物-病原体相互作用以及激素信号转导相关,这些基因可能在从共同祖先分化后,为响应局部环境而发生了动态演化。

图4:石斛属倒位维持了古老的遗传变异
六、TE推动石斛演化和多样性形成
文章发现石斛属基因组大小差异主要由TE扩增导致,平均77.4%的石斛属基因通过WGD事件以及各种单基因复制模式进行了复制,作者注意到转座复制基因的数量存在动态变化可能与石斛属中多样的TE扩增有关。相较于由WGD产生的基因,单基因复制产生的基因进化速率更高,此外重复基因对之间的表达差异也受到TE的影响。
LTR反转录转座子在石斛物种分化后的500万年间经历了显著扩增。LTR的爆发可能有助于石斛属物种适应局部环境变化。
作者进一步为17个石斛属基因组构建了超级泛TE文库,鉴定出44,837个非冗余TE家族,将其分为三类:核心TE、可变TE和独特TE,独特TE家族的数量远多于核心TE家族,这表明石斛属基因组中TE类型存在显著差异。独特TE对靶基因表达的抑制作用远强于核心TE。与携带核心TE的基因相比,携带独特TE的Ks更小、Ka/Ks更高且Pfam注释率更低,这表明独特TE对基因的高进化速率和分化有影响。TE在石斛属进化过程中,推动了基因组和基因的多样性。
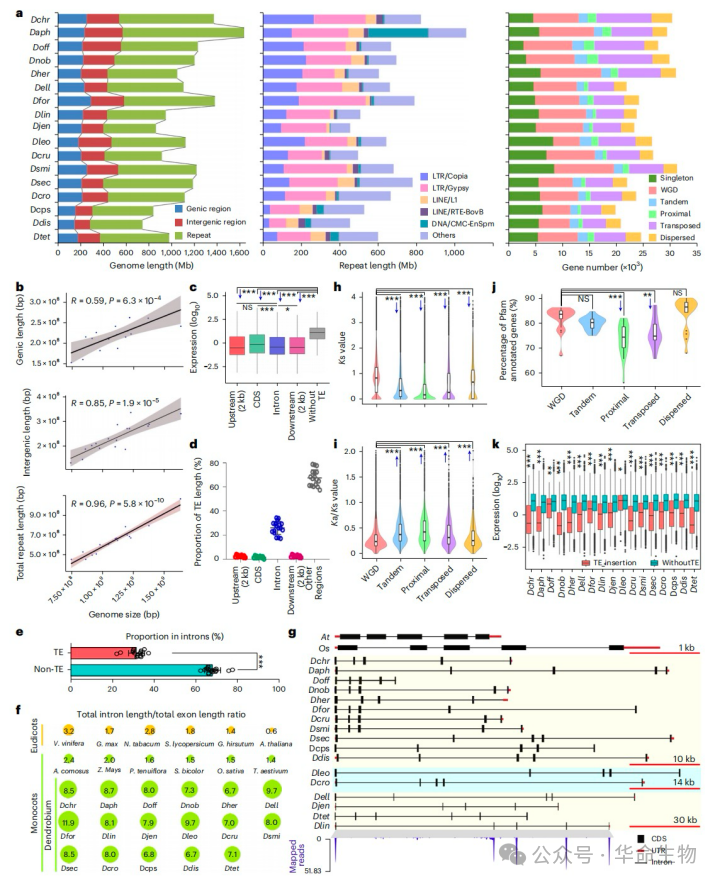
图5:石斛属TE研究
七、MADS-box与FT-like基因演化
文章最后详细探讨了石斛属MADS-box基因的分类、功能及其在植物发育中的关键作用,特别是II型基因在花器官确定和开花时间调控中的重要性。通过分析不同石斛物种中AGL12同源基因的功能与进化,揭示了其在根系发育和适应性进化中的重要作用。此外,文章还深入研究了MADS-box基因簇的复制机制及其在石斛属中的进化历程,特别是AP3-like基因的快速进化和FT-like基因的扩展,为理解兰花独特的花形态特征和适应性进化提供了重要见解。

图6:石斛属MADS-box与FT-like基因演化历史
结语
本研究通过17个石斛样本的染色体级别基因组组装,揭示了石斛属系统发育关系和遗传多样性。文章发现石斛属在从兰科其他支系分化后保留了高度保守的基因和古老的遗传信息,如核心基因和保守非编码序列(CNS)。石斛属北方和南方支系,分别具有独特的分子特征,如北方支系中LTR-RT的特异性扩展。此外,石斛属通过基因家族扩张和染色体重排适应多样生境,尽管石斛属物种未经历特异的WGD事件,但近期TE的爆发显著推动了基因组进化和多样性。TE的活跃还促进了新基因的产生和基因表达的分化,对石斛的生殖发育和生态成功至关重要。这些发现增进了对兰科生物学的理解,并突显了TE在石斛属基因组多样性和功能进化中的关键作用。
参考文献
"Pangenome graphs and their applications in biodiversity gPangeneric genome analyses reveal the evolution and diversity of the orchid genus Dendrobium“ Nature Plants;https://doi.org/10.1038/s41477-024-01902-w
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


