Nature重磅丨中国科学家成功解析食蟹猴T2T基因组
食蟹猴和猕猴是生物医学研究中非常重要的非人灵长类模型,广泛应用于人类疾病研究。然而,它们的基因组复杂性和种间遗传差异尚未完全阐明。近日,来自中国上海交通大学的毛亚飞老师研究团队和中国科学院脑科学研究所的孙强老师研究团队一起,在著名顶刊《Nature》上发表了题为“Integrated analysis of the complete sequence of a macaque genome”的论文,通过构建食蟹猴的完整T2T基因组,填补了这一领域的空白,为理解灵长类进化、基因调控机制以及优化生物医学模型提供了重要资源。

一、T2T基因组组装与注释
研究团队通过孤雌生殖技术获得了一个稳定的二倍体食蟹猴胚胎干细胞系(MFAS82-1),并利用53× PacBio HiFi、77× ONT ultra-long和Hi-C等多种测序数据,经过多轮组装和手动调整,最终完成了食蟹猴端粒到端粒(T2T)完整的基因组组装(T2T-MFASv1.1),填补了此前基因组中的空白区域,基因组大小为3.06Gb,NG50达到了162.13Mb,QV值更是达到71.27。
注释预测了21,119个蛋白质编码基因和12,077个非编码基因,并发现了204个新基因,包含112个新发现的融合基因。通过全长转录组测序,发现这112个融合基因在食蟹猴体内均有表达。进一步发现了795个在食蟹猴和猕猴中差异存在的选择性剪接事件,包含577个基因,作者验证了其中110个外显子跳跃事件。例如,PNPO基因的第5号外显子在食蟹猴中普遍发生选择性剪切,而在猕猴中则未发现。
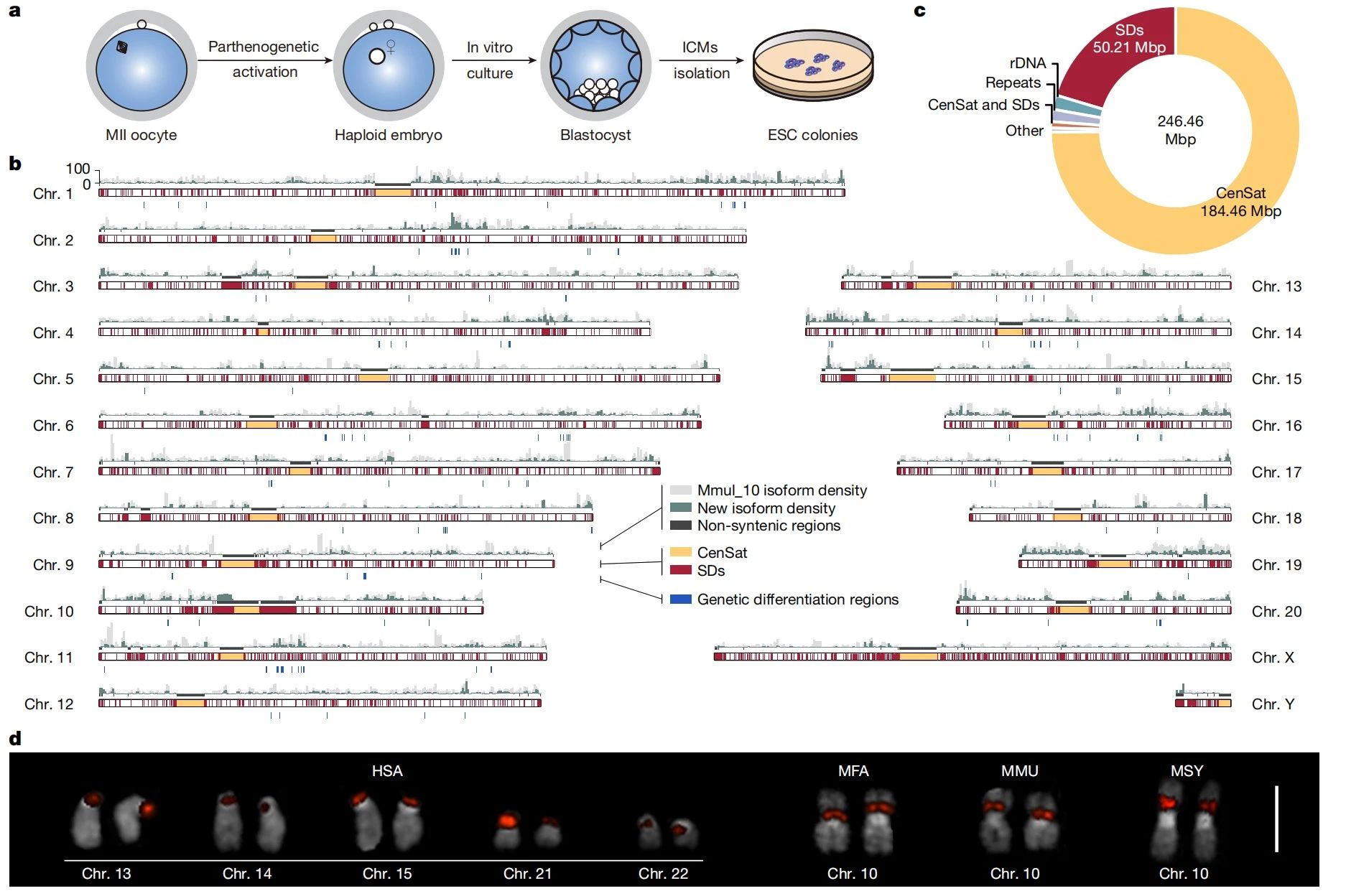
图1:完整的食蟹猴T2T基因组
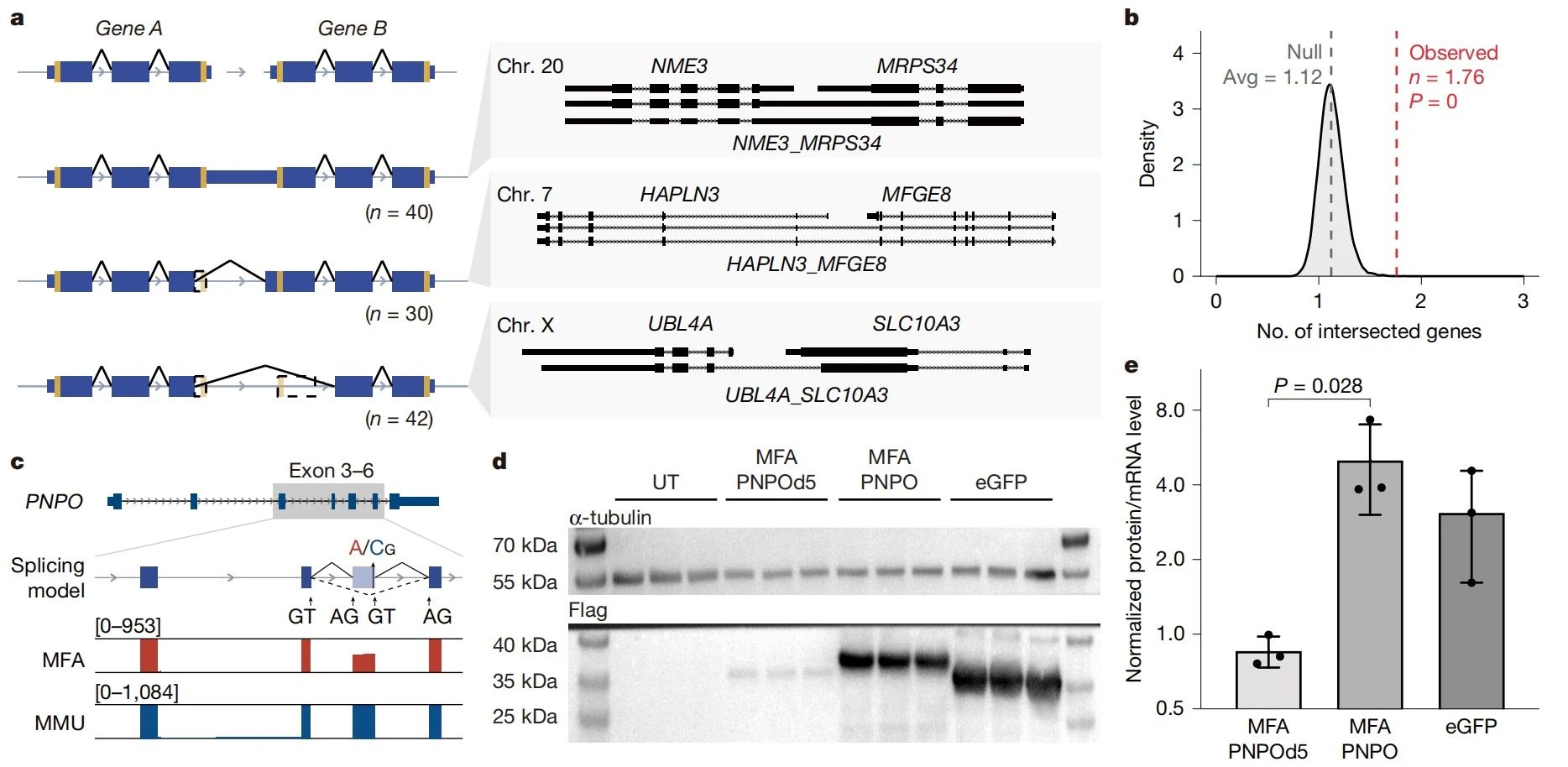
图2:融合基因和可变剪切位点
二、基因组复杂区域解析
研究成功组装了约4.56 Mbp的rDNA区域,并通过微滴数字PCR实验验证了这一结果。与人类基因组不同,食蟹猴基因组中仅存在一个rDNA区域,FISH技术对多种猕猴物实验结果也均支持这一结论。
通过T2T基因组成功解析了122.51 Mbp的节段重复区域(SDs)。其中,65.23 Mbp为染色体间节段重复,101.97 Mbp为染色体内节段重复,与人类基因组(T2T-CHM13v2.0,227.36 Mbp)相比,食蟹猴基因组的SDs减少了104.85 Mbp。
食蟹猴的着丝粒由不同的α-卫星超染色体家族组成,此前尚未在全基因组水平上得到全面解析。本研究发现,食蟹猴的着丝粒主要由SF7衍生的序列构成,两侧则分布着较小的、更古老的SF8-SF13片段,这些区域的核心部分由S1S2a和S1S2b二聚体组成,外围则分布着更为多样化的小片段。

图3:食蟹猴着丝粒和SDs等区域解析及PNPO可变剪切验证
三、食蟹猴与猕猴遗传差异
为更好的理解食蟹猴(MFA)和猕猴(MMU)的遗传差异,作者通过三代测序分别构建了食蟹猴和猕猴的泛基因组图谱,发现了总长度约为240Mb的370个SDR热点区域。这些热点区域包含17个基因,每个基因至少有四个拷贝。同时,通过基因分型和拷贝数变异分析,鉴定出26个在MFA和MMU之间表现出显著变异的基因。
为了更深入地了解MFA和MMU的固定和多态性遗传变异,作者利用94个MFA个体、67个CMMU个体和88个IMMU个体的WGS数据,通过PanGenie鉴定了558,157个SNVs、76,997个InDels和2,797个SVs。
作者采用综合方法(π多样性、FST和XP-EHH)鉴定了约16.76 Mbp的基因组区域,这些区域在MFA-CMMU和MFA-IMMU之间均表现出遗传分化。相关基因涉及包括糖原代谢、神经系统发育以及干细胞多能性调控等多种生物学功能。这些新发现的固定遗传差异可能由选择或遗传漂变塑造,是未来功能实验的重要资源,可用于阐明MFA和MMU之间的各种表型差异。
此外作者结合泛基因组和其他数据,解析了MFA和MMU之间存在的倒位和着丝粒物种特异性差异。
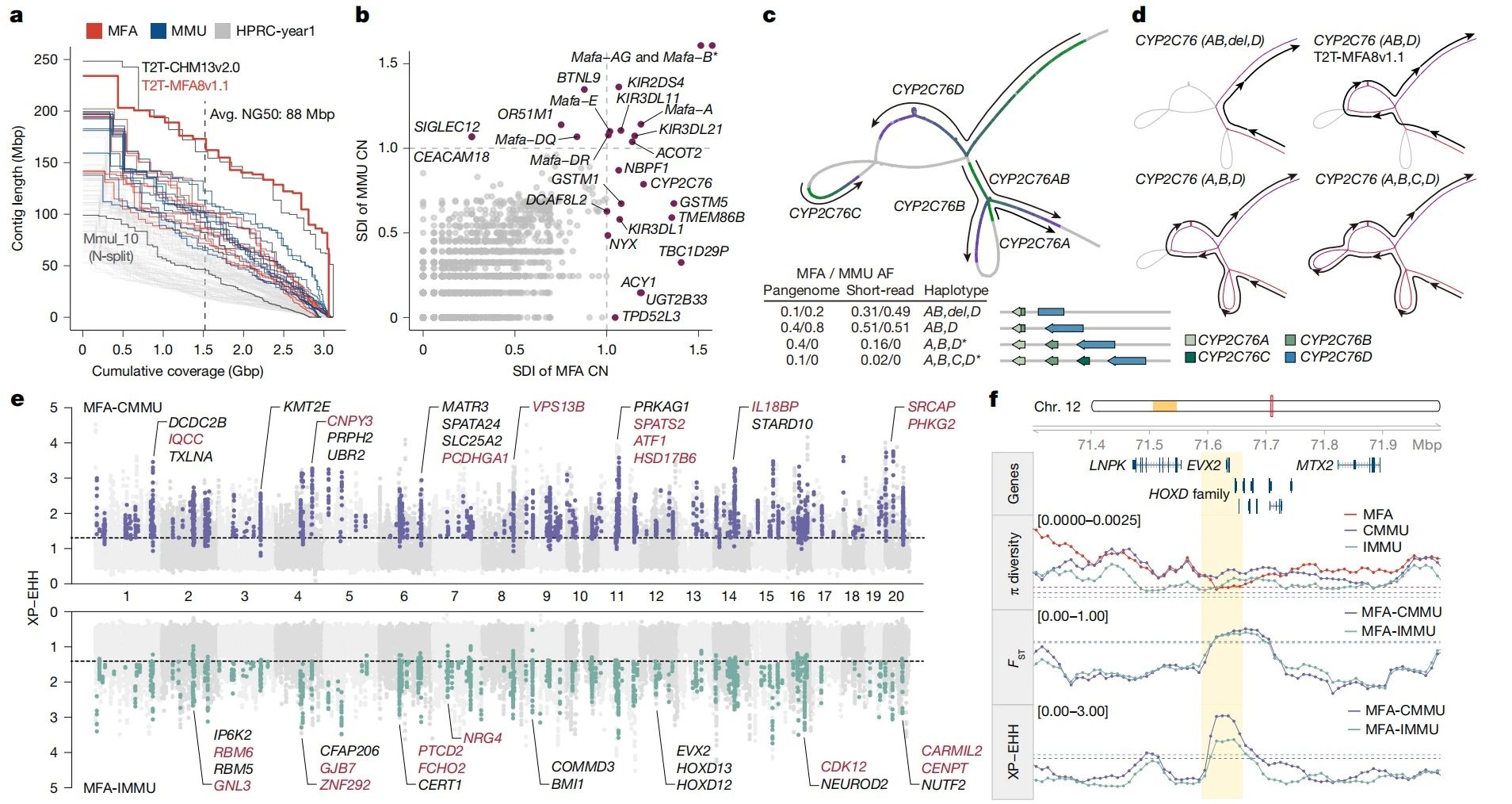
图4:20个单倍型解析的猕猴和食蟹猴基因组解析遗传差异区域
四、人类与猕猴基因组完整比较
作者通过T2T-CHM13v2.0和T2T-MFA8v1.1共线性分析和手动校正鉴定了171个同源区域,这些同源区域在灵长类进化过程中被11个着丝粒重定位、78个倒位和4个染色体内易位分隔。其中21个大尺度基因组重排是本文首次报道。作者选择了三个区域进行FISH验证并证实。在93个大尺度基因组重排及其断点±500 kbp区域内,共包含1,462个蛋白质编码基因。其中,482个基因在人类和猕猴大脑中的表达存在显著差异。除了组织水平的差异表达基因分析外,作者还结合了人类和猕猴脑组织的scRNA-seq数据,鉴定出414个基因,这些基因不仅在大脑组织中表达水平不同,还在细胞类型中表现出不同的表达模式。
在这些基因中,FOLH1和FOLH1B尤其引人注目,在人类中,FOLH1B基因含有一段1,393 bp的特异性缺失,人类的FOLH1在少突胶质细胞中表现出高且特异的表达,而FOLH1B在脑细胞中的表达极低。通过单细胞表观基因组数据和ENCODE数据库,作者证实了FOLH1B缺失区域中存在三个调控元件,这一特异性缺失很可能改变了FOLH1B在人类中的转录。此外,FOLH1在不同脑细胞类型中的表达模式在人类和猕猴之间存在显著差异。进一步分析了Hi-C数据,发现猕猴FOLH1基因组区域内存在两个潜在的sub-TADs。相比之下,人类FOLH1区域中未观察到明显的sub-TADs。这些差异表明,FOLH1在人类和猕猴之间的易位重复可能驱动了不同细胞类型中的基因表达变异,进而改变了染色质结构和调控景观。
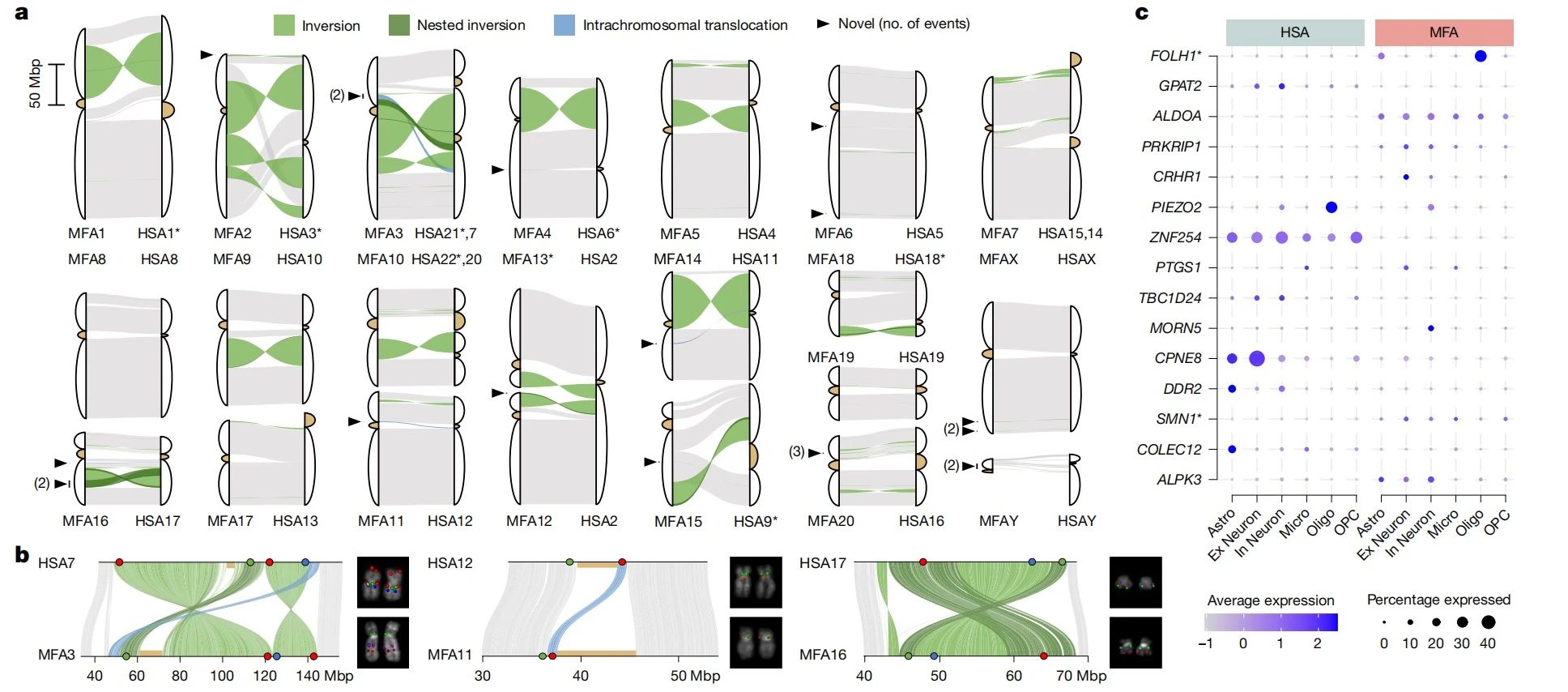
图5:人和食蟹猴基因组差异比较
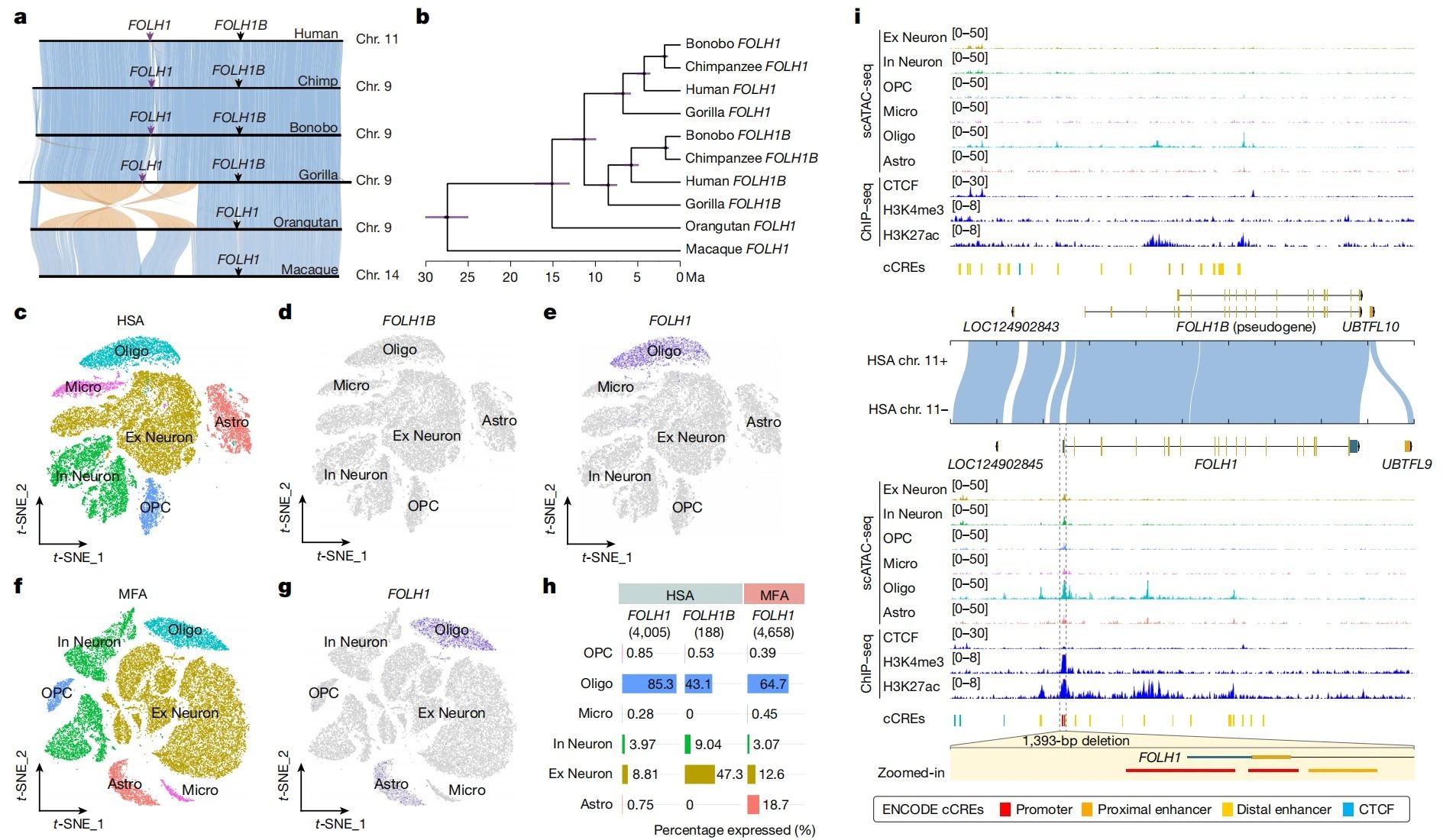
图6:人和食蟹猴FOLH1和FOLH1B差异演化
结语
本文通过完整的食蟹猴T2T基因组denovo组装和多组学分析,不仅为食蟹猴的生物医学研究提供了坚实的遗传基础,还深化了对灵长类进化、基因组结构变异和基因调控机制的理解。研究结果具有重要的科学和应用价值,为未来的人类疾病研究和灵长类进化研究提供了新的方向和资源。
参考文献
Zhang, S., Xu, N., Fu, L. et al. Integrated analysis of the complete sequence of a macaque genome. Nature (2025). https://doi.org/10.1038/s41586-025-08596-w
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


