Nature重磅丨六个猿类单倍型T2T基因组
华命生物目前已成功完成50+物种的T2T基因组组装,物种涵盖动物、植物、昆虫及同源和异源多倍体等疑难物种,已有多个合作项目在顶级期刊发表和接收,欢迎有需要的老师垂询。联系方式:18371456025。
自2001年人类基因组首次测序完成以来,获取高质量猿类基因组序列始终是人类遗传学界的核心任务。这些基因组的完整测序对重建人类基因组每个碱基对的演化历史至关重要。但由于猿类基因组的高度重复性,完整T2T组装始终未能实现。现有参考基因组在诸多复杂基因组区域仍缺乏序列完整解析。
2025年4月9日,来自全球的多个科学家团队合作,在顶级学术期刊Nature上发表了“Complete sequencing of ape genomes”的研究论文,文章首次完成了六种大猿物种完整单倍型分型的T2T基因组组装,尽管部分rDNA区和部分最大着丝粒区域仍待完善,但这些基因组在质量上显著超越已发表的大猿参考基因组,与T2T-CHM13人类参考基因组质量相当。

一、六个猿类单倍型T2T基因组组装
本研究聚焦六个猿类雄性样本(黑猩猩、倭黑猩猩、大猩猩、婆罗洲猩猩、苏门答腊猩猩和长臂猿),旨在同步解析Y染色体序列,实现各物种全染色体的完整覆盖。所有样本通过生成高深度的PacBio HiFi(平均深度90×)与ONT 超长数据(平均深度136.4×,其中UL>100 kb至少30×)以及Hi-C和NGS测序数据,采用Verkko混合组装算法和后续人工校正,将74%的(290条中215条)染色体实现端粒至端粒(T2T)的完整组装,整体评估显示99.2-99.9%基因组实现完整精确组装,包括异染色质区域。该精度指标与T2T-CHM13v1.1参考基因组相当。
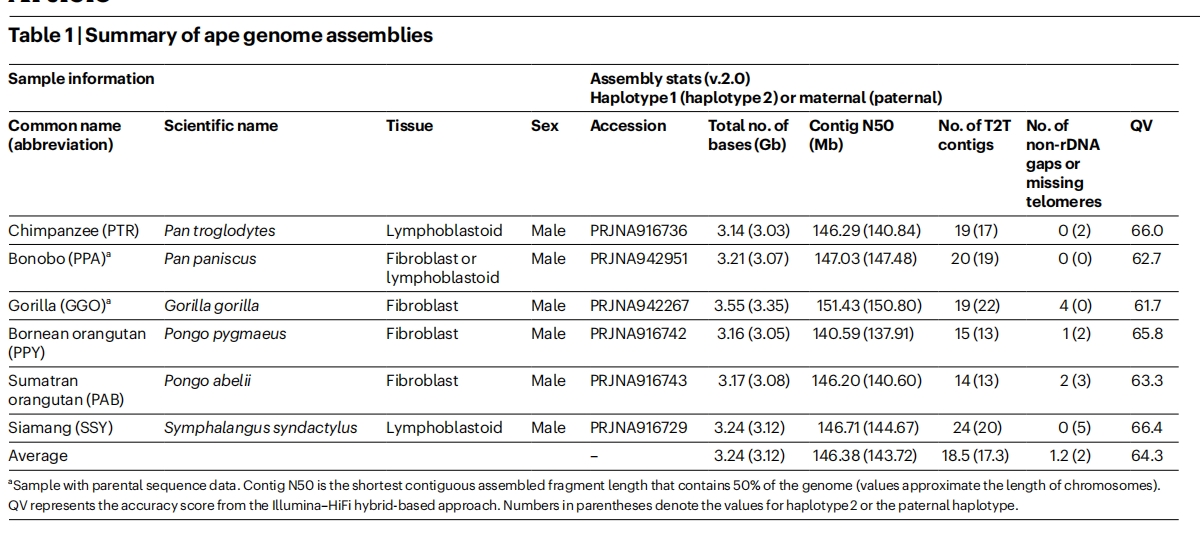
表1:猿类单倍型T2T基因组组装结果汇总
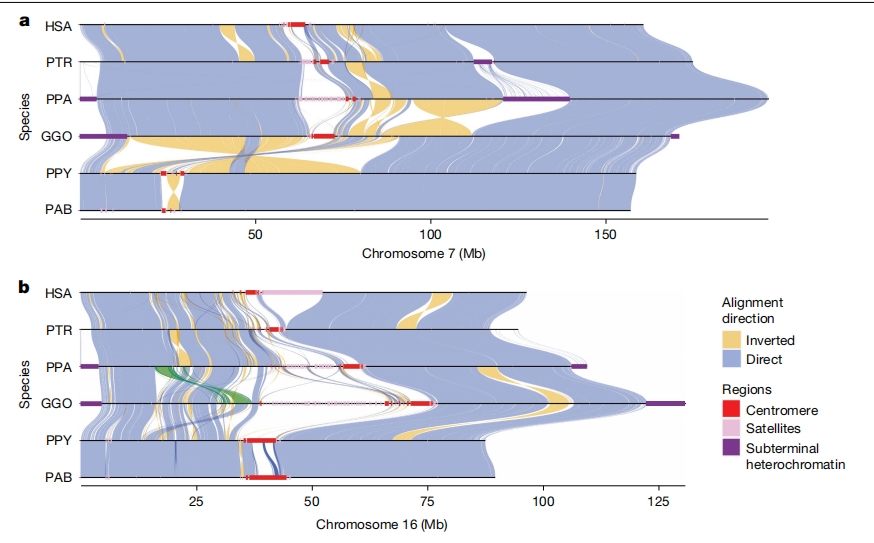
图1:5种大猿完整的16号染色体与HSA(a)和HSA(b)的比较
二、演化分化与自然选择
全基因组对比揭示猿类物种间序列分化程度显著高于既往估算,通过系统解析所有结构分化区域(SDRs),发现各猿类谱系平均存在327 Mb(占基因组10%)的SVs,涵盖着丝粒、近端着丝粒短臂、亚末端异染色质等预期区域,以及大规模重排断点处富集的基因丰富区。
基于可靠比对区段,作者构建猿类物种演化树,推算出关键分化时间:人-黑猩猩分化时间为550-630万年前,非洲大猿共同祖先分化时间1060-1090万年前,红毛猩猩分化时间1820-1960万年前。
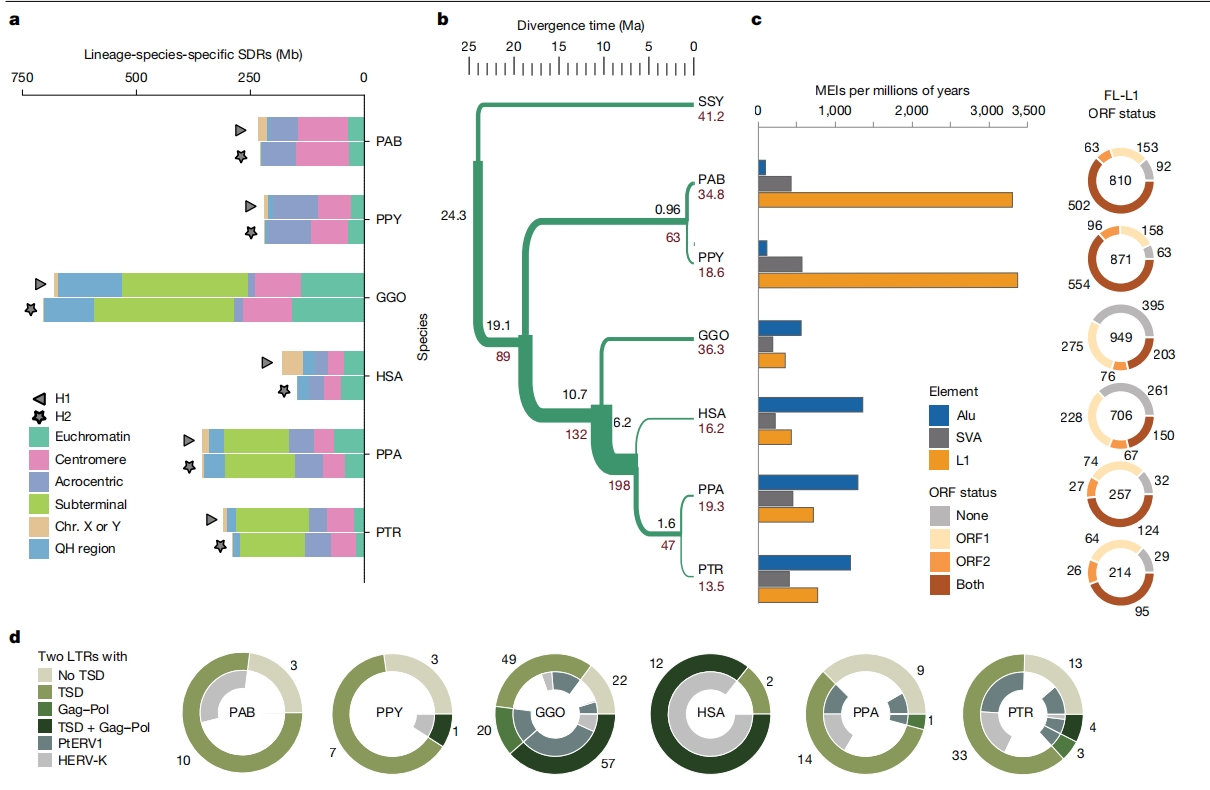
图2:演化分析与重复序列
三、跨物种基因差异解析
在NHP T2T基因组中鉴定出3.3%-6.4%的蛋白编码基因存在人类注释未收录的新转录模型。其中68.6%与SDs相关,且均获Iso-Seq转录本支持。非SD重叠基因拷贝中,73%转录模型发生>50%序列改变,27%丧失翻译潜能。此外,2.1%-5.2%转录本呈现NHP特异性新型剪切变体,均获Iso-Seq数据支持。
四、重复序列全景注释
作者系统分析了猿类基因组中所有高拷贝重复序列及其分布特征。分析显示,常染色体重复序列占比53.2%-58.0%,该比例显著低于性染色体。相较于以前的基因组组装,T2T组装重复序列总量增加286-706Mb。大猩猩、黑猩猩、倭黑猩猩与合趾猴基因组呈现显著卫星DNA扩增,主要源于谱系特异性卫星序列与VNTR的亚末端异染色质累积。
五、卫星DNA谱系分化
卫星序列变异度最高,婆罗洲红毛猩猩卫星含量最低(4.9%,159.2 Mb),大猩猩最高(13.0%,462.5 Mb)。首次鉴定出159个新型卫星单体,各基因组新增注释0.5-7.1 Mb序列。其中大猩猩基因组中发现36 bp新型VNTR_148重复单元,总长3.8 Mb,显著高于倭黑猩猩(841.9 kb)与黑猩猩(55.9 kb)。
六、转座元件动态演化
作者通过多重序列比对精确定义了LINE、Alu、ERV及SVA等逆转座子的全长与截短型插入事件。红毛猩猩展现最高LINE-1(L1)转座活性,但Alu元件失活,表明其基因组环境更利于L1转座优势竞争。
在保留靶点重复序列及完整Gag、Pol结构域的全长ERV中,大猩猩特异性ERV含量最高(57个),其次为人类(12个)与黑猩猩(4个)。系统分析显示,PtERV与HERV-K为主要功能型ERV,而大猩猩、人类、黑猩猩及倭黑猩猩基因组中更多为降解型ERV。
七、免疫球蛋白与T细胞受体基因座解析
基于T2T完整基因组,首次系统性解析大猿免疫相关复杂结构区域。聚焦9个免疫应答与抗原呈递关键区域,揭示其受复杂突变过程或选择压力驱动的演化特征。
免疫球蛋白(Ig)与T细胞受体(TCR)基因家族通过体细胞重组实现抗原识别多样性。分析显示各物种单体倍平均包含:IGHV 60个、IGKV 36个、IGLV 33个、TRAV/TRDV 46个、TRBV 54个、TRGV 8个功能性基因。免疫球蛋白基因座单体型间长度差异达33%,显著高于TCR基因座(10%),提示免疫球蛋白因无需受MHC分子互作限制,演化速率更快。
所有IGH单倍型均存在跨基因串联重复扩增,大猩猩IGK单体型显示长程重排,倭黑猩猩IGL单体型存在1.4 Mb倒位。这些大尺度变异对应物种特异性基因簇。IGH基因座物种特异性基因数量最多,且>10 kb的SDs密度显著高于其他基因座,表明其基因组结构是驱动种内免疫多样性的关键因素。
八、MHC基因座全景解析
基于T2T完整单倍型基因组,作者系统性构建12个猿类MHC区域(4-5 Mb)单倍型注释图谱。跨物种比较揭示:相较人类基因组,非人灵长类(NHP)MHC区域平均存在328 kb缺失与422 kb插入事件。MHC I类基因在NHP种内及种间结构变异显著高于II类基因,合趾猴MHC区域分化尤为突出,功能性MHC-C基因座缺失,代之以人类中仅为假基因的MHC-J样基因座。红毛猩猩两个物种均显示MHC-A与MHC-B基因扩增,且单倍型特异性保留/丢失MHC-C。
除DRB基因座外,NHP MHC II类基因座高度保守。作者整合已发表黑猩猩、大猩猩等MHC单倍型数据,证实MHC基因结构变异广泛存在,符合长期平衡选择理论。
九、SV与染色体重排全景解析
基于T2T完整基因组,作者系统性验证26种人猿核型分化标志性大尺度染色体重排事件。通过断点精细解析,首次揭示多例复杂连续倒位演化模式:
大猩猩18号染色体演化重塑:4.8 Mb基因富集区经三次连续倒位易位至下游12.5 Mb,纠正既往"简单倒位"错误分类;
红毛猩猩2号染色体复杂重构:婆罗洲猩猩经历三次倒位+着丝粒重定位(ENC),苏门答腊猩猩四次倒位+ENC。
另外新鉴定1,140个种间倒位(>10 kb),其中522个为首次发现。基因型分析显示纯合倒位632例,多态性倒位508例,功能关联性416例倒位断点涉及编码基因。63.5%倒位断点显著富集人类同源SDs。
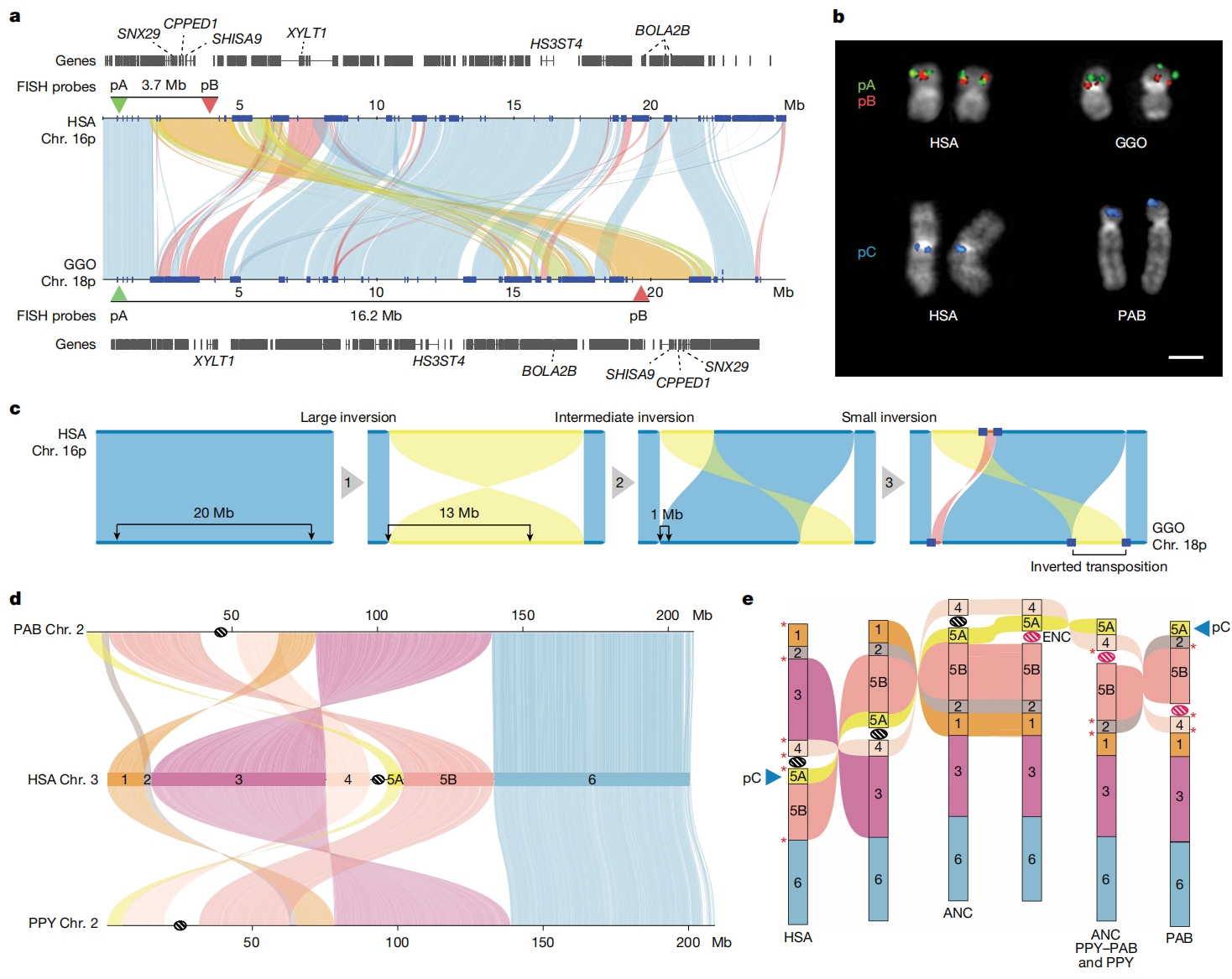
图3:SV与染色体重排演化
十、NORs动态演化
人科动物NORs染色体归属呈现显著种间差异。例如人类15号染色体(HSA15)为NOR+,而黑猩猩/倭黑猩猩中为NOR-;HSA18在黑猩猩中为NOR+,人类中为NOR-。
十一、近端着丝粒染色体短臂特征解析
近端着丝粒染色体短臂呈现"卫星序列富集-编码基因贫乏"特征,NORs定位于常染色体短臂末端或Y染色体长臂末端,但大猩猩HSA2A等染色体虽具短臂异染色质,但无NORs且携带多个蛋白编码基因,揭示异染色质与NORs非绝对关联。短臂区域共线性快速退化,红毛猩猩近端着丝粒染色体以HSat3和α卫星主导,其他大猿呈现均衡卫星组成。
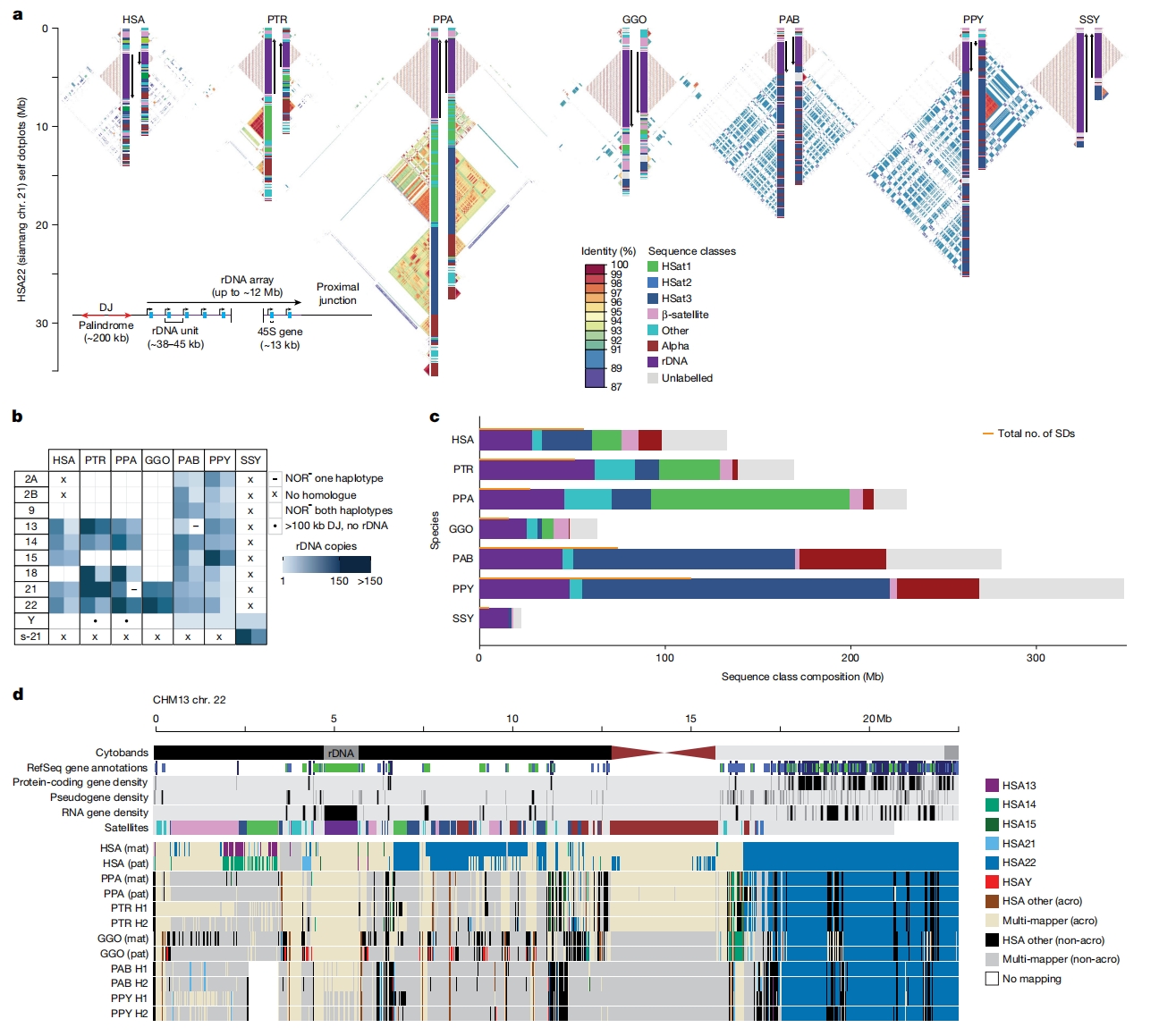
图4:近端着丝粒染色体短臂特征和序列组成
十二、着丝粒卫星序列的演化
通过对五种NHP T2T基因组分析,作者在五种NHP中成功解析了230个可能着丝粒中的227个连续着丝粒。这些着丝粒均由串联重复的α-卫星DNA构成,并进一步组织成属于一个或多个α-卫星超染色体家族(SFs)的高阶重复单元(HORs)。在特定灵长类谱系中,不同SFs呈现高频分布特征,例如猩猩中的SF5和大猩猩中的SF3。
十三、着丝粒谱系特异性特征
针对完整组装的着丝粒,作者发现了各灵长类物种的独特特征:
倭黑猩猩:其α-卫星HOR序列平均长度仅为人类的0.65倍,且比姐妹种黑猩猩短26%。进一步分析显示,倭黑猩猩的HOR阵列长度呈双峰分布——48个着丝粒中,27个平均长度为110 kb,其余21个平均达3.6 Mb。尽管倭黑猩猩与黑猩猩分化时间较近,后者未出现“微型着丝粒”序列。
黑猩猩:其HOR序列长度稳定为人类的0.86倍,通常由单一HOR阵列构成,两侧伴有短片段 divergent HORs和单体序列,这些区域与转座元件交错分布并延伸至染色体短臂(p臂)和长臂(q臂)。
大猩猩:HOR序列平均长度为人类的1.58倍,且由序列内部高度一致但与相邻区域差异显著的 punctuated HORs组成,外围存在较大的单体α-卫星过渡区。大猩猩着丝粒单倍型变异显著:23对HOR阵列中,30.4%(7对)的尺寸差异>1.5倍,39.1%(9对)的同源序列分歧度>5%。
红毛猩猩:婆罗洲猩猩和苏门答腊猩猩的HOR序列平均分别为人类的1.52倍和2.11倍,存在多个 divergent HORs簇。
十四、近缘物种比较揭示快速演化
通过对比黑猩猩属(黑猩猩与倭黑猩猩)和猩猩属的着丝粒,我们得以研究较短演化时间尺度下的α-卫星演化。黑猩猩与倭黑猩猩56%的同源着丝粒可追溯的共同祖先序列。该染色体上倭黑猩猩整个HOR阵列与黑猩猩某一结构域相似度达92–99%,但黑猩猩着丝粒还存在另一个占HOR阵列约50%的新结构域(与倭黑猩猩相似度<70%),表明该新HOR亚区是在黑猩猩谱系中形成并扩张的。
分化时间更短的猩猩属(90万年)则显示更高保守性(HORs序列相似度>97%),但仍有约1/5着丝粒存在种特异性HORs片段(图6f)。在HSA4、HSA5等5条染色体上,新形成的猩猩HORs序列表现出>99%的同质性,提示快速更替与均质化过程。
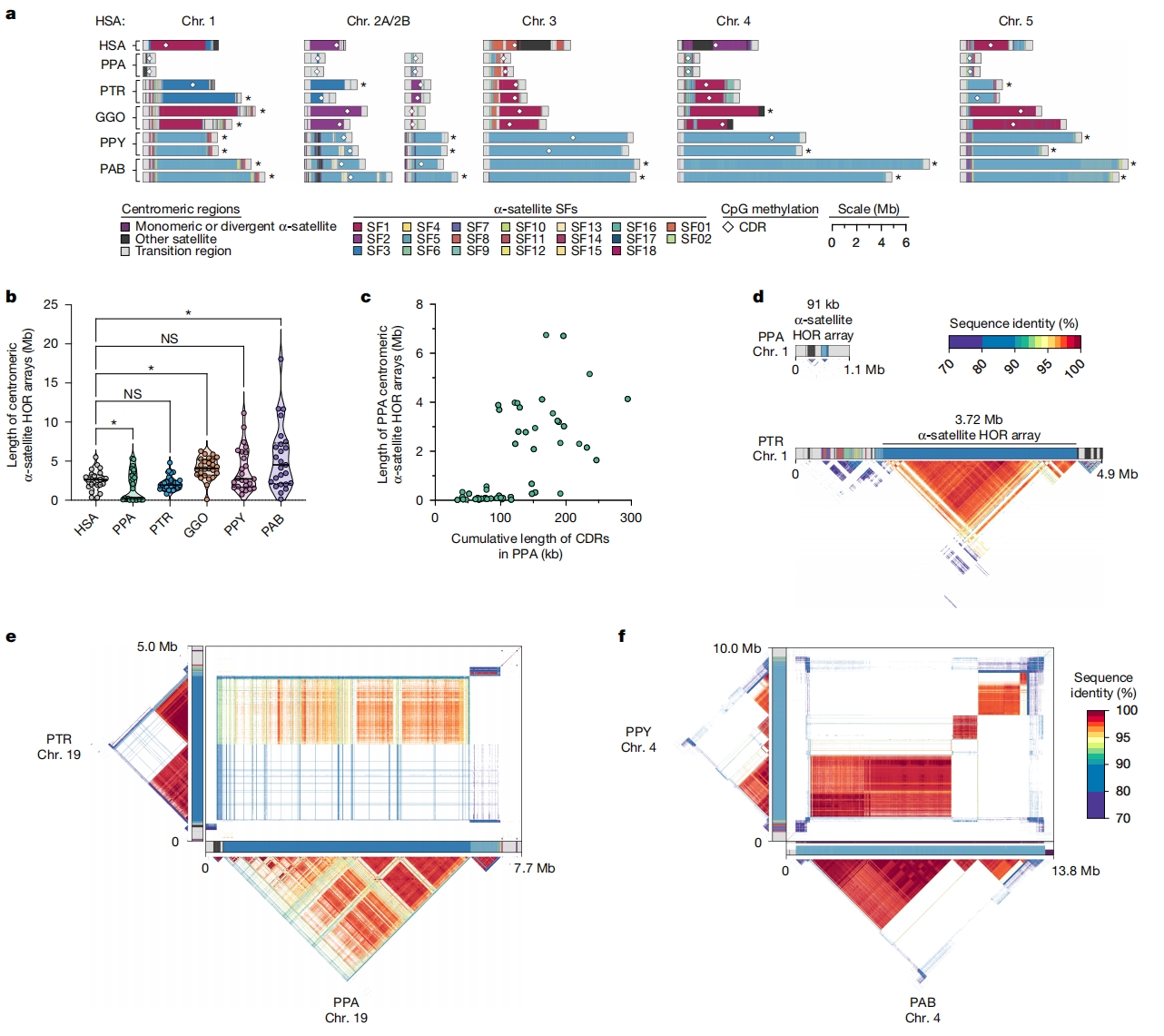
图5:猿类着丝粒卫星序列的组装和解析
十五、近端粒异染色质研究
除着丝粒外,作者首次完成对长臂猿、黑猩猩、倭黑猩猩和大猩猩近端粒异染色质帽的全序列测定与组装。这些区域占基因组比重分别为:黑猩猩270.0 Mb、倭黑猩猩261.6 Mb、大猩猩522.9 Mb(约4–7%),而长臂猿高达642 Mb(10.1%)。这些巨型结构(最长26 Mb)几乎全部由串联重复DNA构成——在黑猩猩属和大猩猩中为32 bp的AT富集卫星序列,在部分长臂猿物种中则为171 bp的α-卫星重复序列。
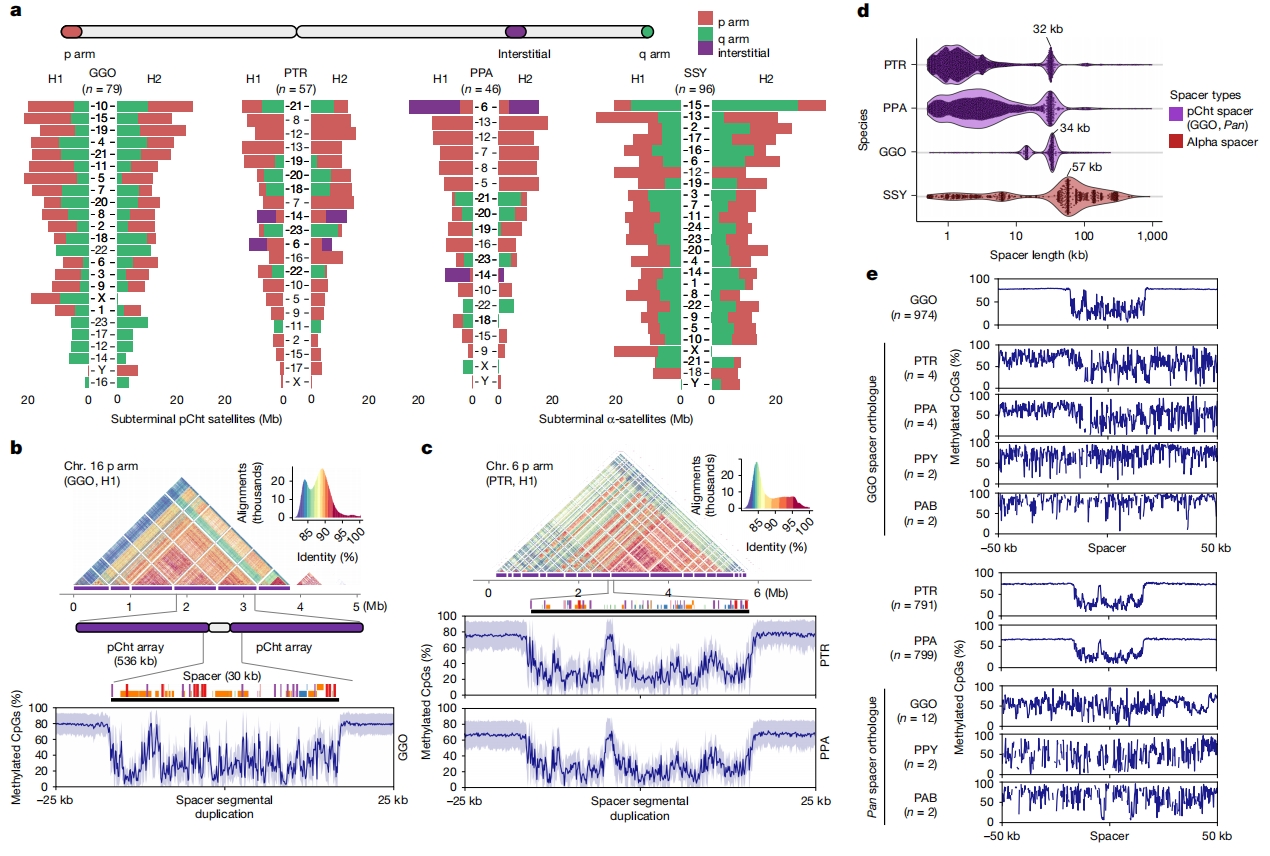
图6:猿类近端粒异染色体区特征
十六、谱系特异性SDs与基因家族演化解析
作者基于构建的T2T基因组,实现了SDs全谱解析,大猿SDs均值为215 Mb,SDs含量显著高于鼠狐猴等非人灵长类。谱系特异性SDs含量排序为:红毛猩猩(100.1 Mb)>大猩猩(69.5 Mb)>人类(41.9 Mb)。
跨单倍型比较揭示30-37% SDs呈多态性,其中苏门答腊红毛猩猩42-50% SDs存在单倍型差异。
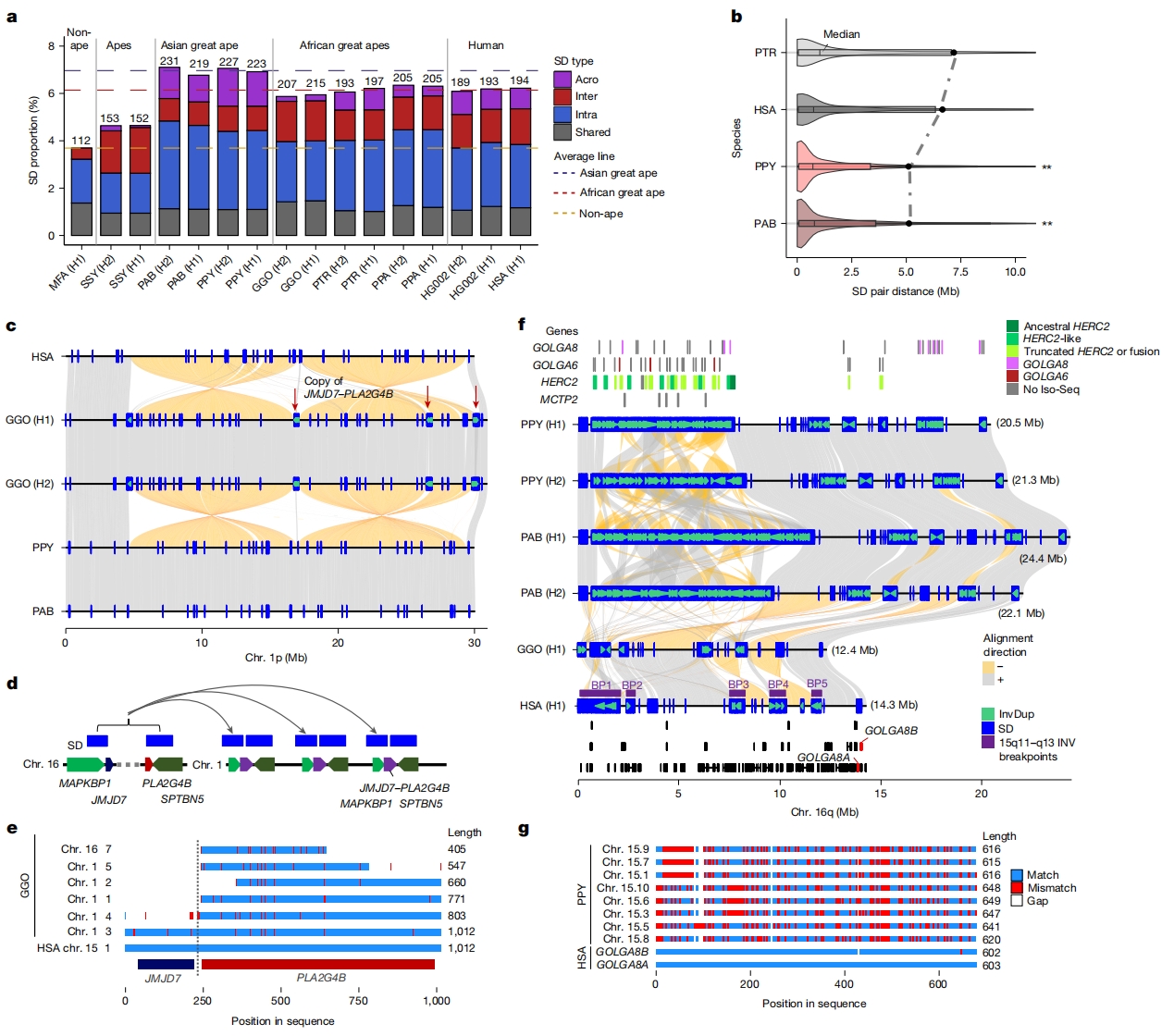
图7:猿类SDs和新基因
研究结论
本文组装了六种猿类(黑猩猩、倭黑猩猩、大猩猩、婆罗洲猩猩、苏门答腊猩猩和长臂猿)的单倍型T2T基因组。通过比较分析,研究了以前未被完整研究的区域,这些区域包括特定谱系的SDs中的新基因家族、着丝粒DNA、亚中着丝粒染色体和亚末端异染色质。这一资源为人类和我们最近的猿类亲属的未来进化研究提供了一个全面的基准。
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


