免费公开丨华命生物改进注释整合工具EviAnn,欢迎大家使用探讨
华命生物目前已成功完成50+物种的T2T基因组组装,物种涵盖动物、植物、昆虫及同源和异源多倍体等疑难物种,已有多个合作项目在顶级期刊发表和接收,欢迎有需要的老师垂询。联系方式:18371456025。
第一部分
传统的注释整合工具, BRAKER3、MAKER3 和 EVM等软件都需要多方证据的支持,如从头预测,同源预测和转录本预测,准备这些数据花费时间长,步骤繁琐,且不稳定。最近,Steven Salzberg 研究团队公布了最新的注释整合工具,EviAnn。地址为:
https://github.com/alekseyzimin/EviAnn_release
经我们测试后,发现了以下问题:
①缺少了从头预测证据,仅依靠转录组和同源预测,最终预测的基因数量往往会低于实际已发表的基因数。
②预测结果不稳定,BUSCO值时高时低,目前在鱼类和哺乳动物中预测良好,但是在昆虫,寄生虫中预测基因效果不佳。
③运行步骤还可继续优化,提高运行速度。
第二部分
对此,华命生物的生信分析团队对EviAnn原版本进行了代码升级与流程优化,并且免费公开给大家使用,也跟原作者反馈了一些提升的意见,请见原地址的Pull requests,下载地址:
https://github.com/linyuiz/EviAnn_update
目前最新的升级版本为:zgtools-EviAnn v2.0.2-2。相较于原版,我们做了以下优化:
①修复了一些原版本的BUG;
②支持用户添加GFF文件作为整合证据。例如,添加从头预测的结果;
③切分蛋白多线程并行比对,加快同源比对的速度;
④转录组支持单端二代测序比对,且支持并行任务。
⑤去除假基因,且最终的结果只提取最长转录本的结果,同时尽可能保持高质量。
第三部分
01 安装
请在releases界面下载最新的tar.gz文件,按照以下示例安装,注意,EviAnn改版需要安装两个依赖hisat2和seqkit,其他均为编译安装。
wget https://github.com/linyuiz/EviAnn_update/releases/download/v2.02-2/zgtools-EviAnn_2.0.2_v2.tar.gz
tar -xvzf zgtools-EviAnn_*.tar.gz
cd zgtools-EviAnn_*
export LD_LIBRARY_PATH=/usr/lib64:/lib64 #防止ufasta安装失败
./install.sh
mamba install seqkit TransDecoder minimap2 hisat2 #or conda install
EviAnn的依赖项请参考:
https://github.com/alekseyzimin/EviAnn_release?tab=readme-ov-file#dependencies。
注意在改版中,除seqkit和hisat2软件外,用户可能还需安装pv命令,安装方式如下:
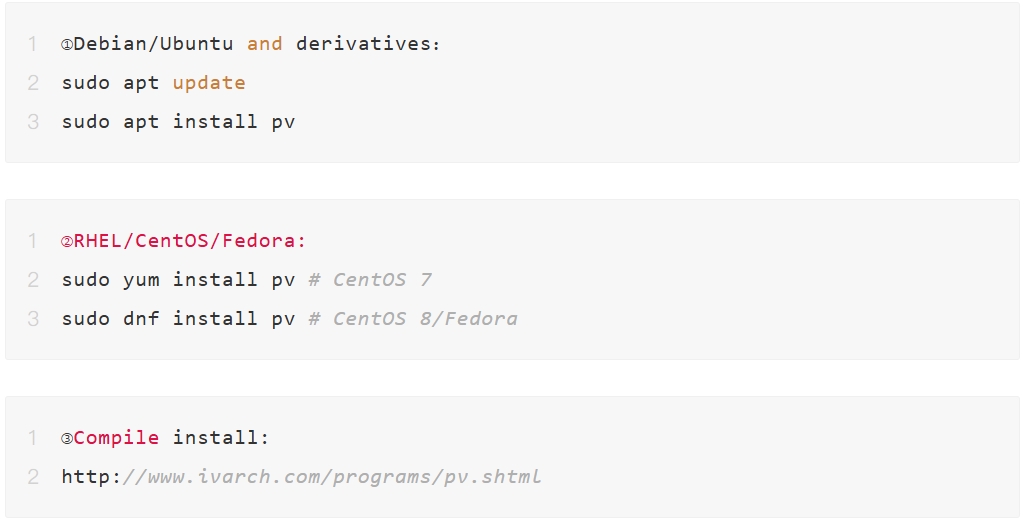
02 数据准备
在使用EviAnn之前你可以按照以下说明进行数据准备:
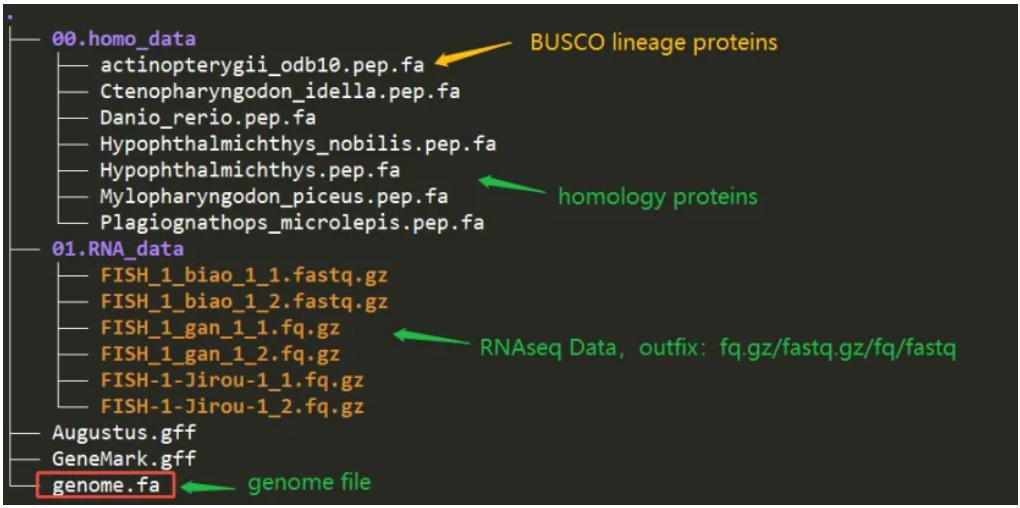
①基因组方面,EviAnn注释不需要经过重复序列屏蔽后的基因组。
②转录组方面,RNA测序数据必须以.fq.gz/.fq或.fastq/.fastq.gz结尾,除了支持双端RNA之外,也支持单端RNA数据。
③同源预测方面,准备近缘物种的蛋白文件,文件必须以.pep.fa结尾。不需要GFF文件,只需要蛋白质文件,且不要求最长转录本提取的蛋白。
对于同源蛋白,建议下载更多序列。一般从5个密切相关物种中选择蛋白质数据就足够了。如果BUSCO完整性分数不够高,您可以扩大密切相关物种的范围,包括更多的蛋白质,甚至高达一百万个蛋白质。此外,您可以使用BUSCO数据库蛋白质作为输入文件,例如将“embryophyta_odb10/ancestral”文件复制为“embryophyta. pep.fa”。
它现在还支持输入一个GFF文件作为从头预测的证据。格式示例如下,请注意必须提供绝对路径:
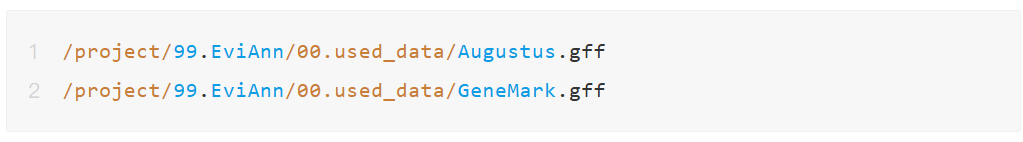
数据准备示例如下图:
03 运行方法
你只需将zgtools软链接到你常用的bin文件夹(如【~/bin】),或者使用绝对路径,并确保【hisat2】和【seqkit】已添加到你的$PATH环境变量中。
Usage:
zgtools EviAnn genome.fa Pep_dir/ RNAseq_dir/ 60 3 Pair_NGS other.gff.list
genome.fa --Genome File
Pep_dir/ --Homo Pep Dir
RNAseq_dir/ --RNAseq Dir
60 --Threads
3 --Parallel Task Num
Pair_NGS --RNAseq Type(Pair_NGS/Single_NGS)
other.gff.list --Other Gff List
Example1:
zgtools EviAnn 00.used_data/genome.fa 00.used_data/00.homo_data/ 00.used_data/01.RNA_data/ 60 3 Pair_NGS denovo.gff.list
Example2:
zgtools EviAnn 00.used_data/genome.fa 00.used_data/00.homo_data/ 00.used_data/01.RNA_data/ 60 3 Single_NGS none
请注意,总线程数是线程数乘以并行任务数,例如:60 x 3 = 180 线程。
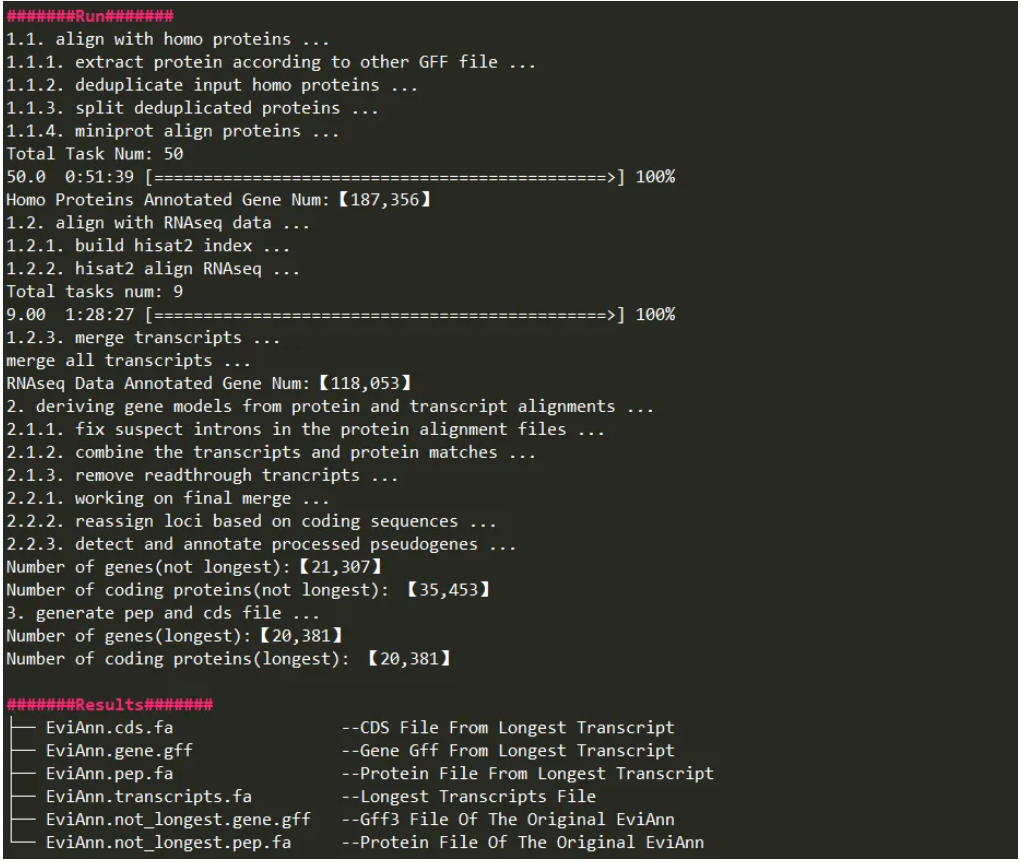
04运行日志
以下是示例命令【zgtools EviAnn genome.fa 00.homo_data/ 01.RNA_data/ 60 3 Pair_NGS other.gff.list】的运行日志:
05结果文件
在output目录下,包含以下结果文件:原版的gff文件和蛋白文件,注意原版的gff包含假基因和lncRNA等标签,此外,还有根据最长转录本提取的cds,gff和蛋白序列文件。

如果遇到任何问题可以在issue部分进行提问,也请大家支持原作者,如果使用了EviAnn也请引用它:Efficient evidence-based genome annotation with EviAnn Aleksey V. Zimin, Daniela Puiu, Mihaela Pertea, James A. Yorke, Steven L. Salzberg bioRxiv 2025.05.07.652745; doi: https://doi.org/10.1101/2025.05.07.652745
如有侵权,请联系工作人员修改或删除。工作人员联系方式:18371456025(微信同)。
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


