NC重磅 | 刘建全团队通过核型动态演变解析十字花科演化
古基因组学通过非基数改变(如倒位、相互易位)或基数改变重排(染色体断裂增加数目/融合减少数目)推演祖先核型。当断点未知时,连锁群减少通常解释为染色体融合,但表型相似的演化事件可能源于不同机制。通过鉴定染色体同线性区块(CLSBs)作为原染色体,并追踪其通过三种重排类型(RCT、EEJ、NCF)的动态演化,可揭示从祖先核型到现代基因组的演化路径。该方法近期在锦葵科古多倍化杂交研究中成功应用,表明祖先核型研究可揭示杂交基因组起源(Sun et al., PNAS, 2024)。
2025年5月19日,刘建全团队在Nature Communications在线发表重磅论文“Chromosome fusions shaped karyotype evolution and evolutionary relationships in the model family Brassicaceae”。利用十字花科33个物种的染色体水平基因组组装数据,通过系统分析三种重排类型,成功重建了两大超族的祖先核型。该方法不仅能独立验证现代芸薹基因组的深层演化关系,还可探究两类古多倍体物种的起源。研究更进一步揭示了染色体融合断点与已知CGBs边界的对应关系。

一、十字花科祖先核型
作者利用WGDI重建了羽衣甘蓝族和芸薹族的祖先核型(ACBK,p=8),鉴定出前四条原染色体。在羽衣甘蓝族中,发现两种祖先核型AKI和tAKI(均p=8),共享ACBK前四条染色体及两条额外原染色体,但7/8号染色体存在易位差异。芸薹族中鉴定出AKII(p=7),含ACBK四条原染色体及三条新原染色体。小果芥(n=6)的基因组分析揭示了AKI5、AKI6和AKII5的保守片段,支持其系统发育位置。
通过外群比较(如Gynandropsis gynandra),确定染色体重排方向性,重建preAKI(p=8)和preAKII(p=7),发现preAKI5与preAKI8融合形成preAKII7,从而确认ACBK剩余四条原染色体。系统发育分析支持ACBK→AKI→tAKI的演化路径,挑战了传统认为tAKI为最原始核型的观点。
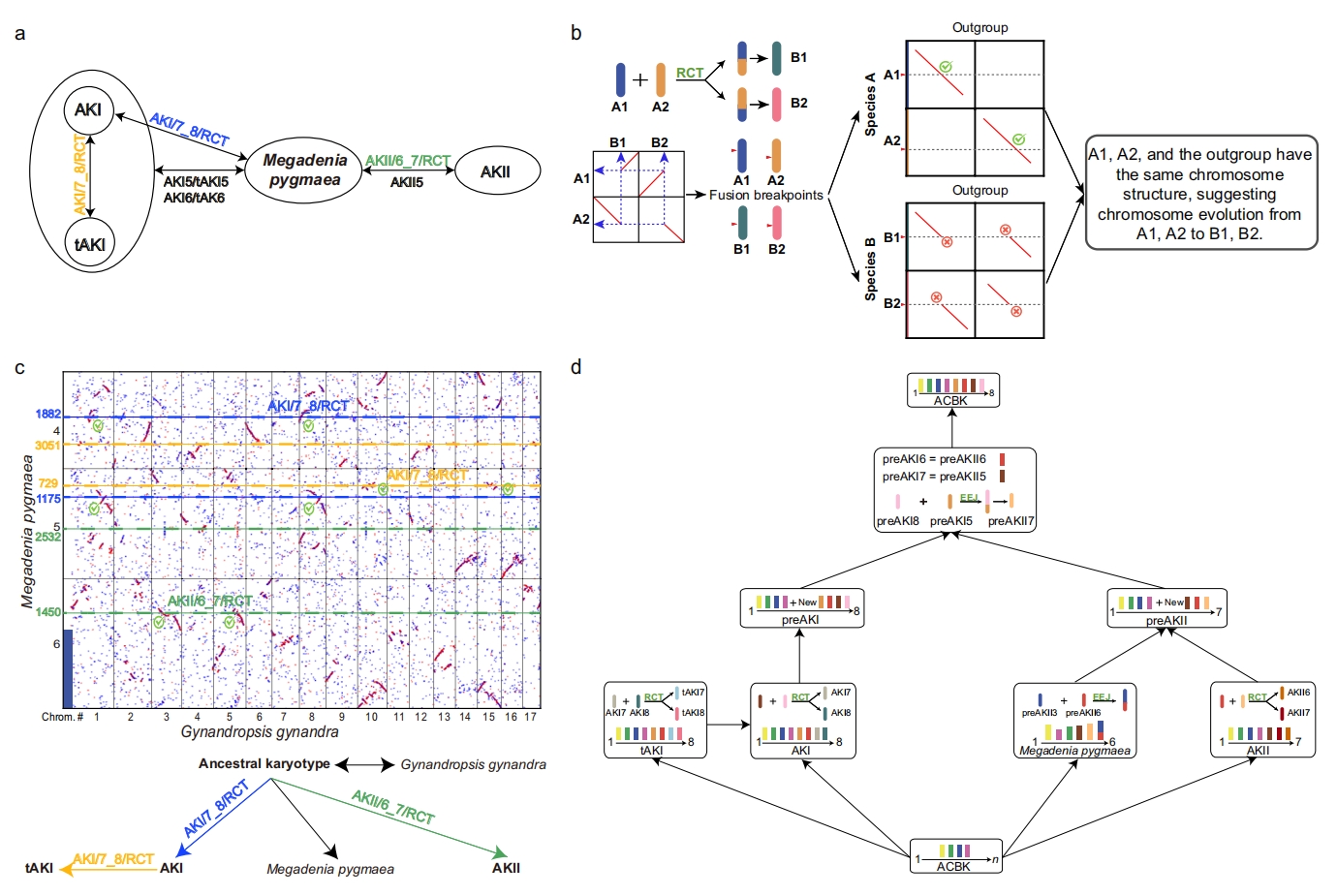
图1:利用外群基因组重建祖先核型的方法与案例
二、十字花科的基数递减现象
作者深入研究了羽衣甘蓝族和芸薹族物种中染色体数目变异与染色体融合事件之间的关系。除碎米荠族物种保留了祖先AKI基因组,其他羽衣甘蓝族物种的基因组都源自通过相互易位(AKI/7_8/RCT)形成的祖先tAKI基因组。三个EEJ事件使拟南芥染色体数从n=8减少到n=5,其他拟南芥属物种仍保留了祖先染色体数目和tAKI基因组。
虽然一些芸薹族物种保留了祖先preAKII核型(p=7),但本研究所分析的四个芸薹族最近共同祖先经历了6号和7号染色体之间的相互易位(preAKII/6_7/RCT),形成了共有的AKII基因组(p=7)。而Heliophilodae中,M. pygmaea(Biscutelleae)经历了preAKII/3_6/EEJ事件,使其染色体数从7减少到6。
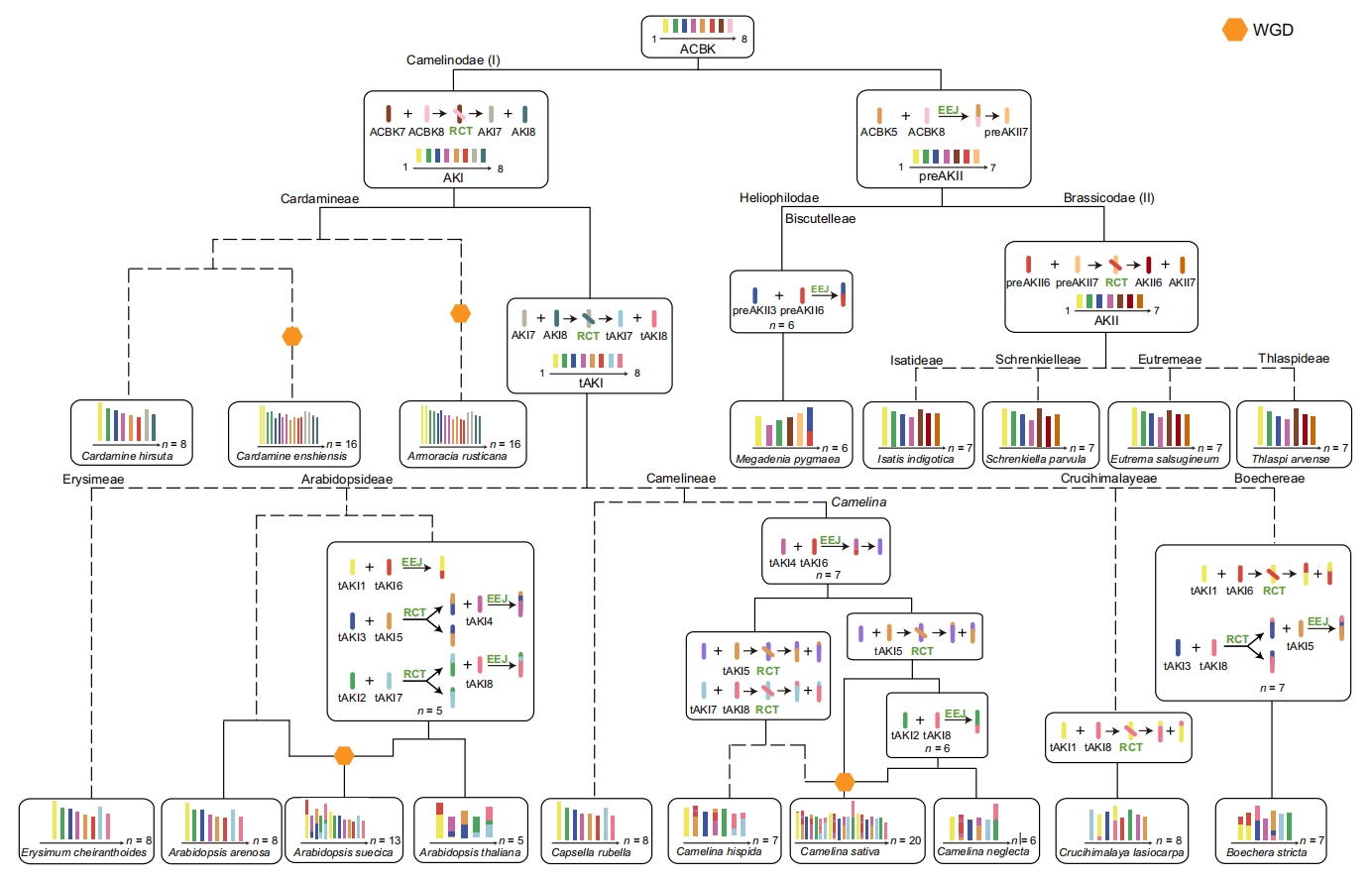
图2:三个十字花亚科超族(芸薹超族、亚麻荠超族和日光超族)的核型演化重建
三、应用共享融合事件确定异源多倍体亲本来源
作者测试了基于独特的基因组特异性染色体融合事件来鉴定异源四倍体Arabidopsis suecica(2n=26)和异源六倍体Camelina sativa(2n=40)的亚基因组的亲本基因组。在四倍体A. suecica基因组(n=13)中,所有三个拟南芥特异的染色体融合都在5染色体亚基因组中被鉴定出来,而基于共线基因构建的亚基因组树,我们得出结论认为A. arenosa最有可能是8染色体亚基因组的供体。
通过融合断点共享分析,作者发现C. sativa的三个亚基因组分别源自C. hispida(n=7)、C. neglecta(n=6)和C. neglecta-like基因组(n=7)的祖先。这一结论得到了亚基因组树的进一步支持。而与先前基于所有染色体结构鉴定亲本基因组的方法不同,作者的方法仅针对推定亲本基因组和杂交基因组中的融合断点。
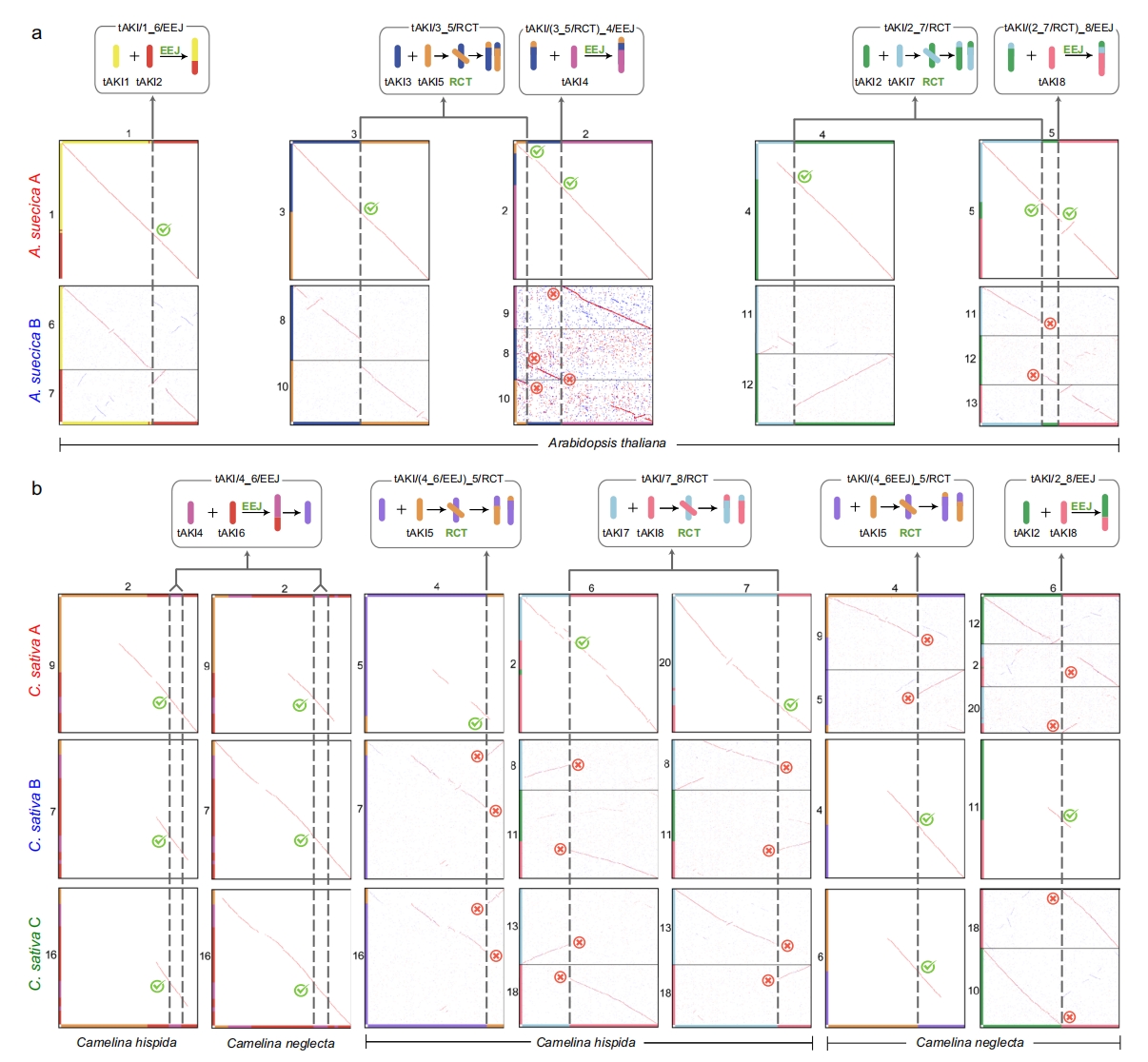
图3:共享的染色体融合事件可用于鉴定异源多倍体基因组中的亚基因组
四、特定染色体重排断点与CGB边界相关
为探究22个保守基因组区段(CGB)边界与融合事件的关联性,作者将共享事件的融合断点(BPs)及CGB边界映射至ACBK核型。对比分析显示除四倍体Lepidium didymum外,所有十字花科科物种均稳定遗传了独特的融合事件,未发现特定融合断点的重复起源现象。而三个外群(Arabodae、Hesperodae和Aethionemeae)均未共享全部融合断点。
对于异源多倍体物种,融合事件的不同拷贝数同样反映了这些杂交基因组的古老起源,相较于传统DNA序列比对方法,这种基于共享染色体融合事件的策略为推断深层进化关系提供了可行且具有前景的新途径。
最后,作者重构了独行菜族演化关系,通过鉴定出两种不同的染色体重排,结果支持了独行菜族与碎米荠族的姐妹群关系。
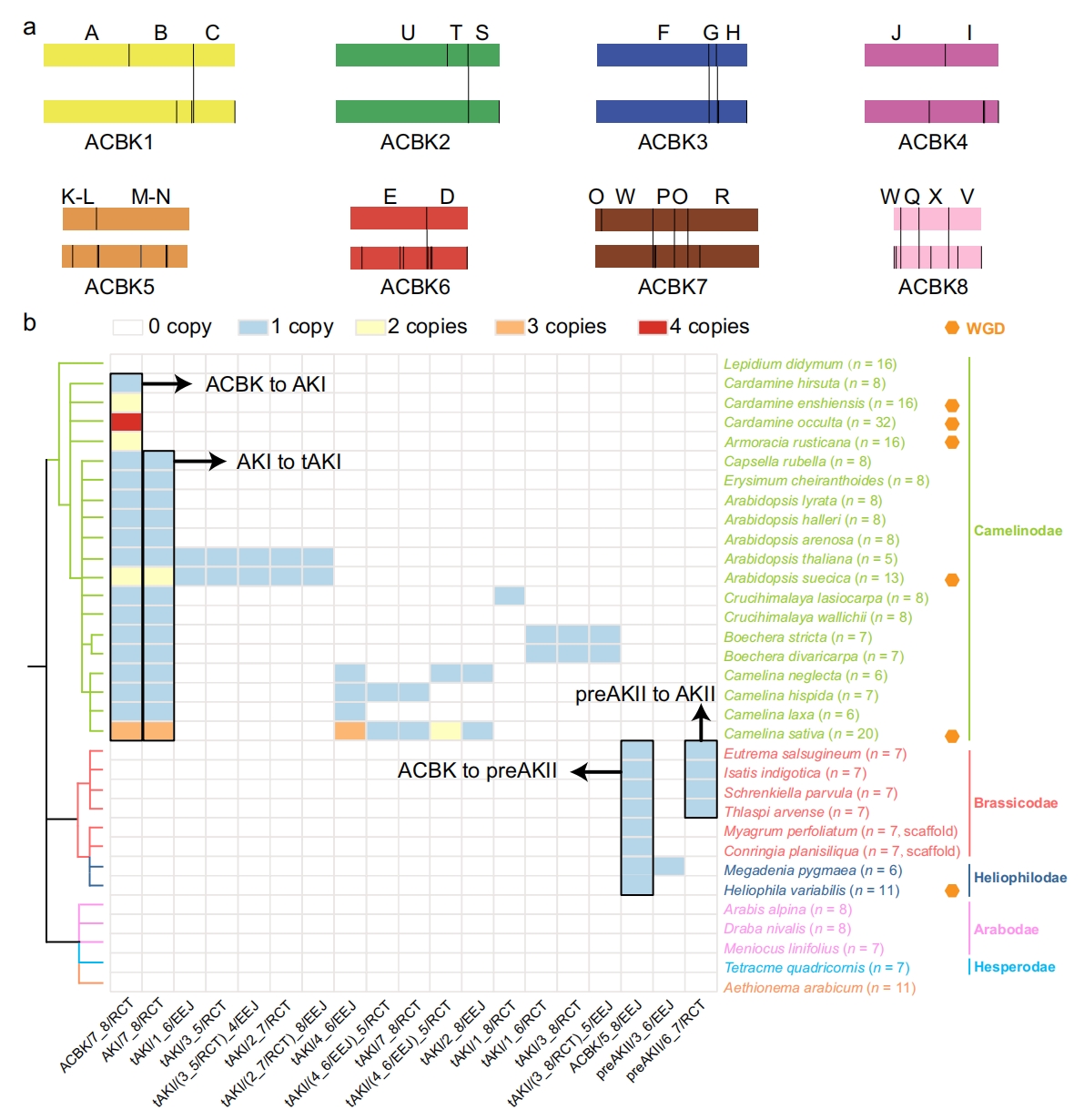
图4:染色体断点与保守基因组区块的关联性及其系统发育信息价值
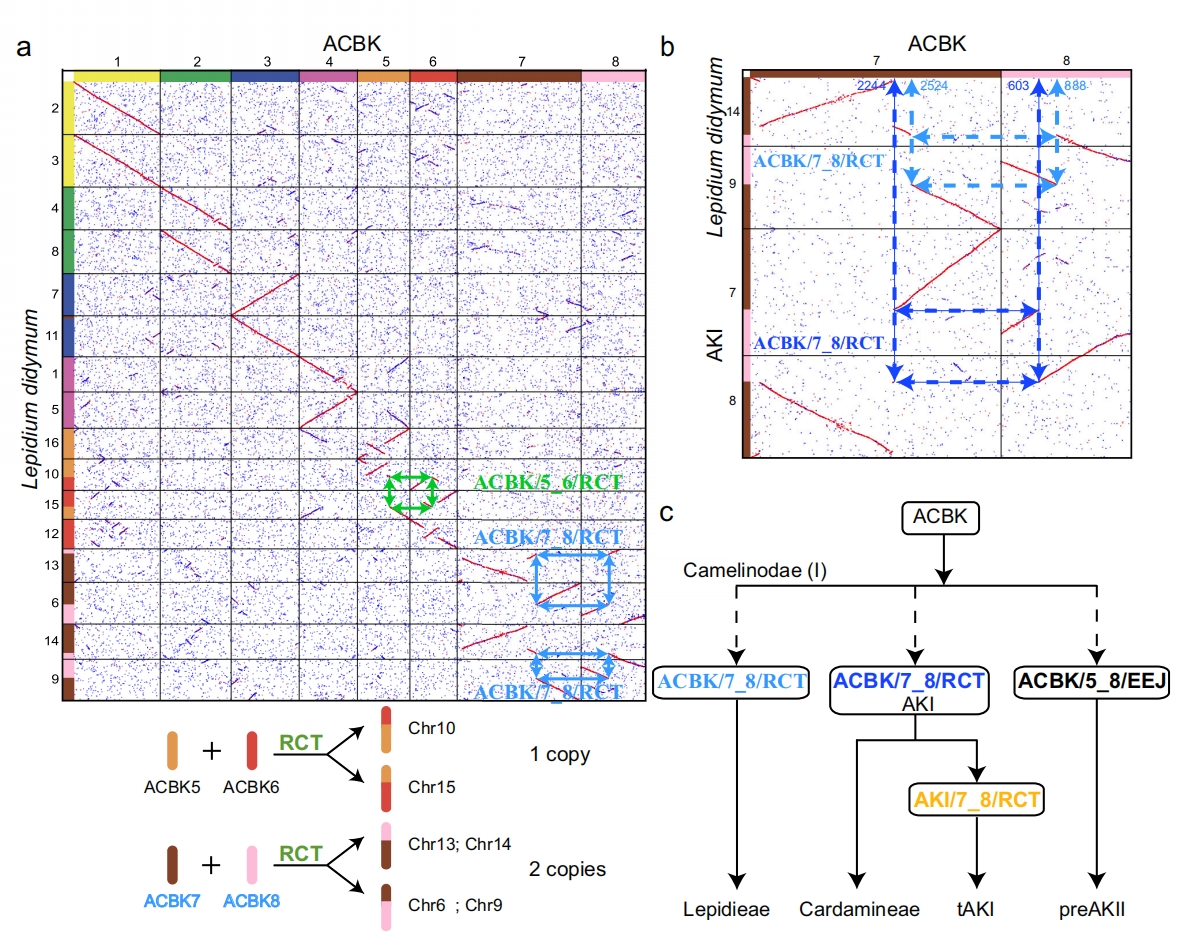
图5:基于古老染色体重排解析独行菜族与碎米荠族的演化关系
结语
不同于传统基于DNA序列的系统发育分析,刘建全教授团队提出了以染色体融合位点作为演化标记的新框架。该研究为解析被子植物核型多态性提供了关键范例。随着测序成本的大幅下降,染色体水平甚至是T2T级别的基因组数据爆发式增长,通过揭示染色体结构变异与物种辐射分化的普遍规律,为理解生命之树的形成提供全新维度。
刘建全教授、马萨里克大学Martin A. Lysak教授和四川大学科研助理孙朋川博士为论文共同通讯作者。
参考文献:
1.Sun, P., Jiao, B., et al. WGDI: A user-friendly toolkit for evolutionary analyses of whole-genome duplications and ancestral karyotypes. Mol. Plant 15, 1841-1851 (2022).
2.Sun, P. et al. Subgenome-aware analyses reveal the genomic consequences of ancient allopolyploid hybridizations throughout the cotton family. Proc. Natl. Acad. Sci. USA 121, e2313921121 (2024).
3.Jiang, X., Hu, Q., Mei, D. et al. Chromosome fusions shaped karyotype evolution and evolutionary relationships in the model family Brassicaceae. Nat Commun 16, 4631 (2025).
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!