Plant Communications丨菠菜和水稻ZH11 T2T基因组两连发
一、菠菜T2T基因组解析Y染色体演化
菠菜(Spinacia oleracea L.)(2n=12),是全球最重要的绿叶蔬菜之一。菠菜属是研究性染色体进化的理想模型,因其雌雄异株、具有XY性别决定系统。近年陆续有菠菜参考基因组构建和发表,但都未达到T2T水平,部分区域仍然存在gaps,这些不完整的基因组组装阻碍了对着丝粒、端粒等复杂基因组区域的探索。近日,中国农业科学院蔬菜花卉所的研究团队,成功组装了菠菜自交系10S15YY雄株的T2T基因组。

作者通过整合了以下多组学数据:90 Gb PacBio HiFi 数据(90×)、65 Gb ONT UL数据(65×,N50=100 kb)、10种组织的30个RNA-seq数据和17 Gb Iso-Seq数据。通过多种组装方法和校正手段,成功组装了自交系10S15 YY雄株的T2T基因组(Sp_YY_v2)。基因组大小为926 Mb,包含12个端粒。通过多种注释手段结合,作者注释了701 Mb重复序列,其中58.63%为LTR反转录转座子。同时预测到28,247个蛋白质编码基因,84%通过Iso-Seq或表达量支持。96.84%基因可在数据库内被注释。
作者还成功获得全长205.40 Mb的Y染色体(chr4),作者解析了Y染色体的完整结构,包括低频重组的着丝粒周缘区(PR)和性连锁区(SLR)。PR区具有更高TE含量和更低基因密度,显著高于非PR区。Y染色体包含24.89 Mb的SLR区,其中10.74 Mb为雄性特异区域(YDR)。在YDR区内鉴定出57个蛋白编码基因,通过71个不同组织样本的基因表达分析,发现一个新的候选雄性决定基因SpRAD,该基因在雄蕊中特异性高表达,且在雌花中完全缺失。
SpRAD基因在栽培菠菜(S. oleracea)及其近缘野生种(S. turkestanica)中保守存在。综合证据表明该基因起源于S. tetrandra分化后(约630万年前)至S. oleracea与S. turkestanica分化前(约80万年前),符合这两物种最近共同祖先中发生的性别决定因子更替事件。
为解析栽培菠菜与近缘野生种的Y染色体演化,作者基于133份S. oleracea和21份S. turkestanica材料,通过SNP检测鉴定出14,248个Y特异SNP。邻接树分析显示:S. oleracea两大谱系分别形成YG1和YG3分支,而S. turkestanica与部分S. oleracea聚为YG2分支。而且在pPAR和SLR检测到三种Y单倍型,其分化程度显著高于重组活跃区(rPAR),符合重组抑制促进遗传变异积累的理论。
作者进一步基于T2T基因组首次揭示菠菜着丝粒特性,通过CRM探针与FISH确认6个着丝粒,长度分别为1.96 Mb(chr5)~4.49 Mb(chr3),其中91%着丝粒序列为TE,Gypsy-LTR占44%,未发现着丝粒特异性串联重复。表观遗传特征显示着丝粒区CHG/CHH甲基化水平均低于侧翼区,而CpG甲基化保持稳定,基因表达量显著低于非着丝粒区。
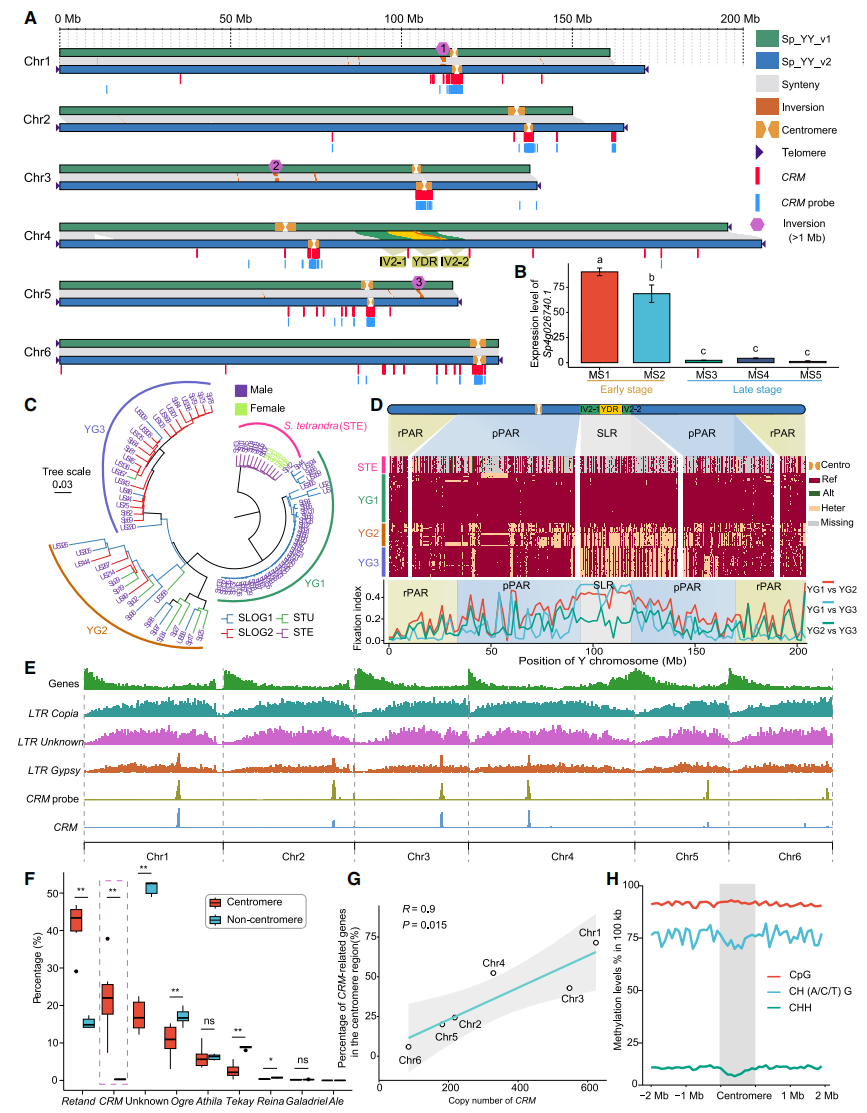
图1:10S15 YY雄株的T2T基因组构建、Y染色体演化研究和着丝粒解析
总结
本研究成功完成首个端粒到端粒(T2T)完整菠菜基因组组装,完整解析了6个着丝粒与12个端粒结构。这一高质量基因组为深入探究着丝粒与Y染色体的演化机制提供了关键基础,将为菠菜功能基因组学研究及遗传改良奠定重要资源基础。
粳稻“中花11”(Zhonghua11,ZH11)因其高转化效率和强适应性成为广泛研究的对象。然而,ZH11基因组组装尚不完整,仍存在25处空缺。近日,中国科学院植物研究所的研究团队,通过多种测序手段,成功完整了ZH11的T2T基因组组装,并通过多种评估方法论证了组装基因组的高质量。

作者通过23.02 Gb PacBio HiFi数据、56.7 Gb ONT ultra-long数据 、24.96 Gb Illumina数据、42.59 Gb Hi-C数据和Bionano光学图谱数据。通过多种组装算法组装、补洞以及端粒延伸等操作,并经过Bionano数据验证,最终获得完整的T2T-ZH11基因组(379.33 Mb)。
作者首先利用Iso-seq、RNA-seq数据及同源蛋白对T2T-ZH11进行注释,共预测出56,948个基因。为验证T2T-ZH11基因组的组装质量并评估其相较于T2T-Nip的改进,进一步整合了147.56 Gb已发表的水稻根部单细胞RNA测序数据及533个品种的全基因组测序数据。
通过Cell Ranger对scRNA-seq数据去重并统计单细胞表达的独特UMI数,结果显示比对率上T2T-ZH11为94.70%,显著高于T2T-Nip的81.3%,而总基因数T2T-ZH11(37,797)较T2T-Nip(36,891)也增加906个。
结合NCBI公开的ATAC-seq、RNA-seq及DNA甲基化数据,并与MSU7.0注释数据对比,发现以T2T-ZH11为参考基因组时,以上数据的比对率、覆盖度及peak识别数均显著提升。
作者基于RiceVarMap数据库中533个水稻品种的400余万SNP,在抽穗期和粒重两个农艺性状中检测到全部关联SNP。通过GWAS比较T2T-ZH11与T2T-Nip基因组,发现关键候选基因(如Hd3a、GW8)的信号位点高度一致,证实了T2T-ZH11组装的可靠性。
最后,作者将T2T-ZH11和T2T-Nip、MSU7.0、羊草、紫花苜蓿等CROPTILLING平台可比基因组数据比较,并将T2T-ZH11整合至CROPTILLING平台(www.croptilling.com/ZH11)。经BUSCO、CEGMA、QV值及LAI值等多项指标评估,T2T-ZH11基因组在准确性与完整性上均达到高标准。
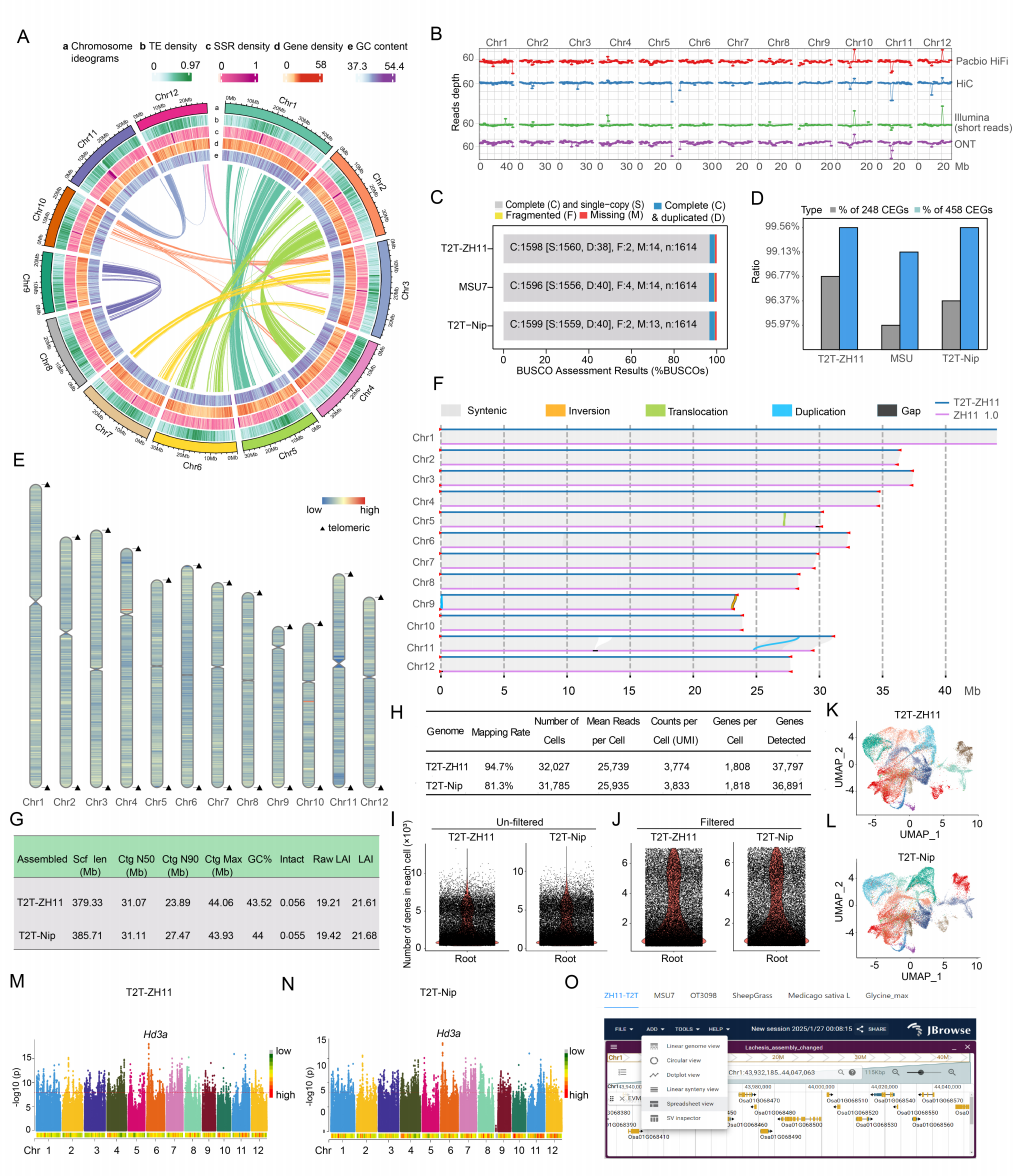
图2:ZH11 T2T基因组构建
总结
本研究成功完成了粳稻中花11(T2T-ZH11)的完整参考基因组组装,该基因组包含379.33 Mb DNA序列、12条完整的染色体。与ZH11-IGDBv1版本相比,填补了25处空缺,使基因组组装长度增加了2.29 Mb。通过整合Iso-seq,基因注释得到优化,预测基因数从37,341增至56,948个,与既往发表的基因组相比,ZH11-T2T在连续性和完整性方面均有显著提升,经多种验证手段证实,T2T-ZH11在准确性和完整性上均达到非常高标准,为水稻的后续育种和相关实验提供了新的高质量参考基因组。
华命生物全面汇总了动植物T2T基因组文章,包含130+已发表的植物T2T基因组文献合集和30+已发表动物T2T基因组文献合集,我们不仅有按照时间顺序排列好的所有文献原文资料包,也贴心的整理了每篇文章的研究物种、测序策略、组装水平、基因组大小等内容,关注华命生物,后台回复关键词:华命交流群,扫码添加工作人员微信,发送单位+名字,管理员验证通过后拉入群聊获取文本全部资料~

华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!