T2T基因组月刊 | 动植物T2T基因组文献更新汇总
6月份华命生物整理了已发表130+篇植物T2T基因组和30+篇动物T2T基因组的文献合集,本期我们更新近期收集的10余篇动植物T2T基因组文献整理。
华命生物目前已成功完成60+物种的T2T基因组组装,物种涵盖动物、植物、昆虫及同源和异源多倍体等疑难物种,已有多个合作项目在顶级期刊发表和接收,欢迎有需要的老师垂询。联系方式:18371456025。

主要研究内容:作者采用多种测序策略对陆地棉(2n=4x=52,AADD)近交系Jin668进行了基因组测序,通过结合387.02 Gb ONT超长数据(169×)和124.88 Gb PacBio HiFi数据(54×),初步组装和Hi-C数据辅助组装后,整个基因组仅有14个缺口。随后综合运用多种补洞策略、端粒修复及重复序列校正,最终获得完整的Jin668 T2T基因组。本研究完成了陆地棉高再生基因型Jin668的T2T完整基因组组装及YZ1的near-T2T基因组组装。通过比较基因组学、染色质可及性及基因表达分析,揭示了Jin668实现高效再生的关键基因调控网络。此外,本研究系统评估了基因型间遗传变异对CRISPR基因组编辑精准度的影响,表明针对特定基因型的高质量基因组组装对确保功能基因研究和分子育种中基因组编辑的准确性具有重要作用。

主要研究内容:作者通过193.19 Gb HiFi测序数据(122×)、654.04 Gb Hi-C数据(414×)和199.24 Gb超长ONT数据(约314×),采用多种组装方法结合和人工填补缺口等方法,成功组装了橡胶树CATAS 7-33-97完整的单倍型分型T2T基因组。本研究成功构建了橡胶树的广泛栽培品种CATAS 7-33-97的首个单倍型分型、端粒到端粒(T2T)完整参考基因组。这一完整基因组不仅揭示了橡胶树复杂基因组的结构特征,还首次明确了单倍型间的结构变异。此外,T2T基因组使橡胶合成相关基因的进化分析成为可能,并揭示了割胶响应背后的橡胶生物合成调控网络。

主要研究内容:作者选取来自浙江省临安县西天目山地区的天目地黄(Rehmannia chingii)TMS11的个体,用于T2T参考基因组组装。本研究整合了 Illumina、PacBio HiFi、Oxford Nanopore(ONT)和 Hi-C测序数据。通过组装和Hi-C数据挂载。最终获得了天目地黄高质量T2T基因组,基因组大小为 1.169 Gb,contig N50长度82.2 Mb。该完整基因组为分析着丝粒和端粒区域、阐明基因组进化与全基因组复制事件,以及鉴定环烯醚萜苷生物合成关键基因提供了重要基础。这些基因组与转录组资源对于地黄的后续功能研究和育种工作具有重大科学价值。

主要研究内容:作者通过整合PacBio HiFi测序数据(81×和113×)、ONT超长测序数据(137×和98×)以及Hi-C数据(101×和153×),通过系统性地采用hifiasm工具对不同数据组合进行从头组装,最终确定了各基因组的最优策略。初始组装后,A17遗留3个gaps,R108存在1个gaps。通过多种补洞策略后,最终成功获得0 gap、端粒到端粒(T2T)的完整基因组A17 v6.0和R108 v3.0。BUSCO完整度评估结果分别为99.19%和99.32%。两个组装均成功识别全部16个端粒。本研究首次完成了蒺藜苜蓿A17与滨海苜蓿R108的端粒到端粒(T2T)完整基因组组装,为豆科植物研究提供了高精度参考基因组。基于CENH3 ChIP-seq测序数据,作者系统解析了两种着丝粒的DNA重复序列特征,这些发现为着丝粒的形成机制和进化动力学研究提供了新视角。

主要研究内容:作者通过23.02 Gb PacBio HiFi数据、56.7 Gb ONT ultra-long数据 、24.96 Gb Illumina数据、42.59 Gb Hi-C数据和Bionano光学图谱数据。通过多种组装算法组装、补洞以及端粒延伸等操作,并经过Bionano数据验证,最终获得完整的T2T-ZH11基因组(379.33 Mb)。本研究成功完成了粳稻中花11(T2T-ZH11)的完整参考基因组组装,该基因组包含379.33 Mb DNA序列、12条完整的染色体。与ZH11-IGDBv1版本相比,填补了25处空缺,使基因组组装长度增加了2.29 Mb。通过整合Iso-seq,基因注释得到优化,预测基因数从37,341增至56,948个,与既往发表的基因组相比,ZH11-T2T在连续性和完整性方面均有显著提升,经多种验证手段证实,T2T-ZH11在准确性和完整性上均达到非常高标准,为水稻的后续育种和相关实验提供了新的高质量参考基因组。

主要研究内容:作者通过整合了以下多组学数据:90 Gb PacBio HiFi 数据(90×)、65 Gb ONT UL数据(65×,N50=100 kb)、10种组织的30个RNA-seq数据和17 Gb Iso-Seq数据。通过多种组装方法和校正手段,成功组装了自交系10S15 YY雄株的T2T基因组(Sp_YY_v2)。基因组大小为926 Mb,包含12个端粒。本研究成功完成首个端粒到端粒(T2T)完整菠菜基因组组装,完整解析了6个着丝粒与12个端粒结构。这一高质量基因组为深入探究着丝粒与Y染色体的演化机制提供了关键基础,将为菠菜功能基因组学研究及遗传改良奠定重要资源基础。

主要研究内容:本研究通过构建毛豆T2T基因组,结合群体测序数据,揭示了毛豆与粒用大豆(GS)的分化选择机制:毛豆以品质和产量为导向,关键基因GmFula、GmPdh1和Dt1分别调控豆荚数、开裂性和生长习性。主导毛豆的F1/P/d单倍型反映了对消费端偏好性状的选择。尽管豆荚开裂率较高,未来通过基因编辑可在保留有利等位基因的同时,规避不良性状。
8.印度亚麻T2T基因组

主要研究内容:研究团队利用PacBio HiFi、Oxford Nanopore(ONT)长读长测序技术和Bionano光学图谱相结合的策略,成功组装了印度亚麻品种T397的T2T级别基因组(495 Mb)。本研究成功构建了首个高质量的印度亚麻品种T2T级别参考基因组,其完整性和连续性远超以往版本。这一里程碑式的成果不仅填补了亚麻基因组研究的关键空白,还为深入解析其重要农艺性状提供了强大的工具。通过整合基因组、转录组、代谢组和群体遗传学数据,研究不仅系统阐明了亚麻富含Omega-3脂肪酸的分子机制,锁定了FAD3和Oleosin等关键基因,还揭示了其花色和种皮颜色的代谢基础,并挖掘出一批具有重要育种价值的优异种质资源。 该T2T基因组将成为全球亚麻研究的基石,极大地推动功能基因的克隆、分子标记辅助选择、基因编辑等现代育种技术的应用,从而加速培育具有更高营养价值、更强抗逆性和更优纤维品质的亚麻新品种。
9.西洋参T2T基因组

研究者通过利用PacBio HiFi测序和ONT超长读长技术组装了西洋参高质量的无间隙T2T基因组,基因组大小为4.33Gb,Contig N50为185.33Mb。与v1.0版本相比,新组装的基因组揭示了大量结构变异(SVs)。本研究首次报道了黄果西洋参品种‘中农洋参2号’(ZN)T2T基因组组装。结合转录组学和代谢组学分析,研究了西洋参不同发育阶段中黄酮类化合物的生物合成及其果实颜色变化的调控机制。研究者对西洋参基因组的系统发育进化的研究,为花青素和黄酮醇的生物合成途径提供了新的见解。这一全面的基因组资源有助于更深入探索黄酮类化合物的多样性,支持遗传改良工作,并提升其在药用植物研究中未来应用的潜力。
10.亚麻T2T基因组

主要研究内容:本研究选取黑龙江省农业科学院提供的油纤兼用型亚麻品种"高丝"为材料,结合Hi-C技术、PacBio HiFi数据和ONT超长读长测序,首次完成亚麻高丝品种的无间隙T2T基因组组装。不仅鉴定了亚麻基因组全部端粒和着丝粒区域,还开展了比较基因组学分析。通过同源比对重新注释了亚麻T2T基因组中的脂肪酸代谢通路基因,并重点解析了脂肪酸去饱和酶(FAD)和硬脂酰-ACP去饱和酶(SAD)基因家族成员。本研究成果填补了亚麻基因组研究空白,为品种选育、遗传改良及分子机制探究提供了理论基础。
1.锦鲤T2T基因组

主要研究内容:研究通过整合PacBio HiFi、ONT超长及Hi-C测序技术,成功构建了两种锦鲤变种(黑鲤和金鲤)端粒到端粒染色体水平的基因组组装。黑鲤、金鲤基因组大小分别为1.55 Gb和1.54 Gb,分别包括50条染色体序列,其中黑鲤基因组检测到100个端粒,金鲤为99个(仅chr48_A10未被完全识别)。该研究成功构建了首个锦鲤T2T基因组,该基因组资源将为深入探究锦鲤体色形成和形态多样性的分子机制奠定重要基础;为皮肤组织的单细胞转录组分析提供基准数据;也为观赏性鲤鱼的标记辅助选择和精准育种研究注入新动力。
2.亚洲冰鱼T2T基因组

主要研究内容:该研究成功组装了亚洲冰鱼的T2T基因组,利用PacBio HiFi长读长、纳米孔超长读长和Hi-C数据,获得了一个大小为380.76 Mb的无间隙基因组,包含28条染色体对应的28个连续序列(contig N50约14 Mb)。该研究提供了亚洲冰鱼的第一个T2T级别参考基因组,显著提升了组装的连续性和准确性,为后续比较基因组学和进化研究奠定了坚实基础。通过大规模群体重测序(231个体),该研究揭示了引入种群因环境压力(如高碱度)导致的遗传分化、多样性损失和自交加剧,这解释了历史上引入失败的原因,并为保护策略(如避免近交)提供了直接证据。发现的适应性基因(如离子结合相关基因)不仅增进了对冰鱼环境耐受机制的理解,还可应用于其他鱼类盐碱适应研究。此外,该研究的基因组资源(NCBI登录号PRJNA1065125)和群体数据支持全球鱼类保护倡议,有助于制定针对性的渔业管理政策,促进生物多样性维持和可持续资源利用。
如何获取本文资料包?
关注华命生物,后台回复关键词:华命交流群,扫码添加工作人员微信,发送单位+名字,管理员验证通过后拉入群聊。群公告中有对应的下载链接。科研群中还有更多科研工具分享,我们会不定期更新文章和科研工具哦!

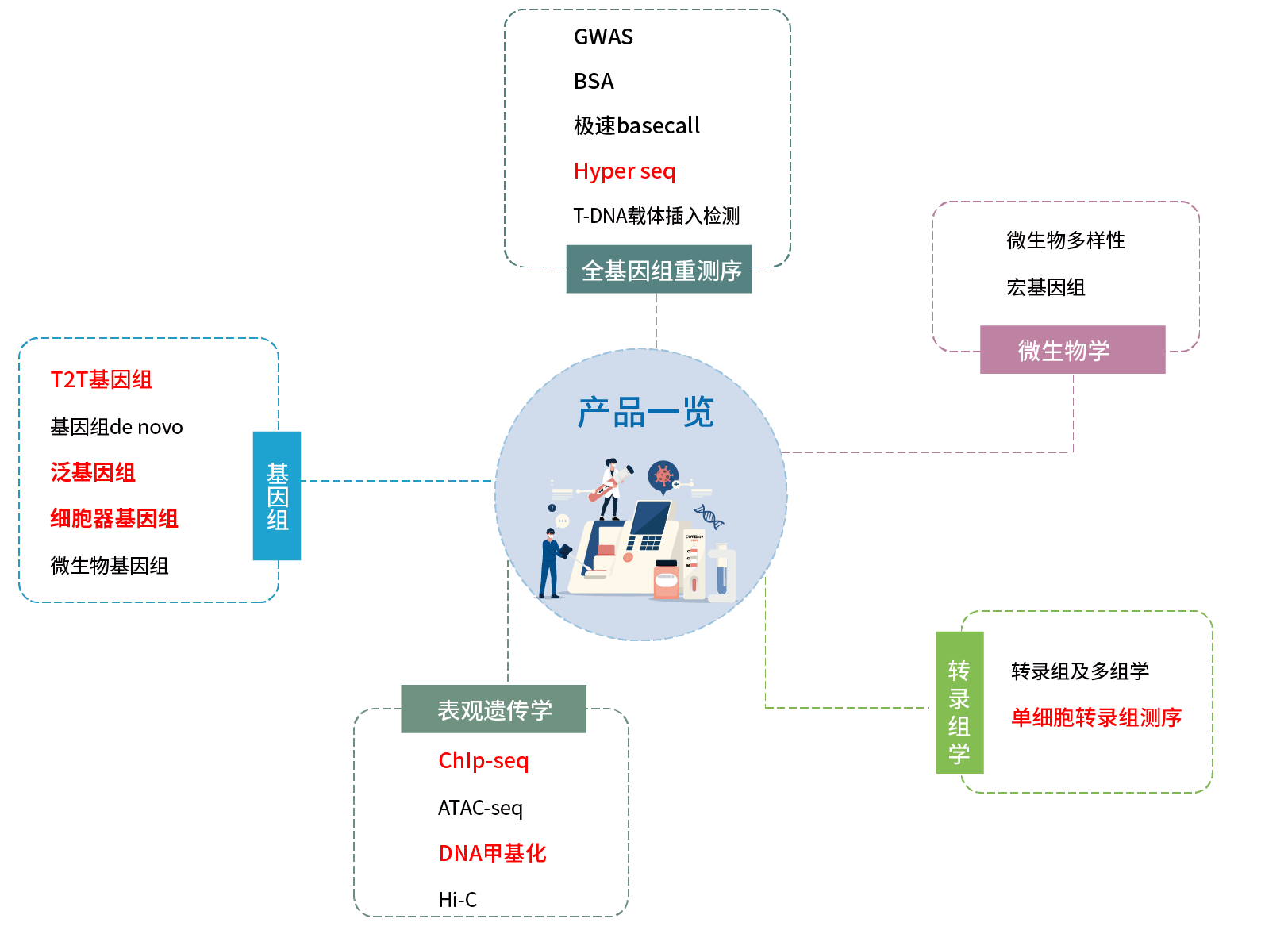
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!