JGG精选 | T2T基因组解码萝卜抽薹
萝卜(Raphanus sativus L.)是全球广泛栽培的重要十字花科根菜作物,其可食用的根、叶及芽富含多种营养。然而,由低温强光引发的提前抽薹会严重影响其产量与品质。从育种角度看,控制抽薹开花时间既关乎经济效益(避免早抽薹减产),又能加速育种进程(利用早花特性)。抽薹开花是受多基因和环境因素共同调控的数量性状,解析其遗传机制对精准育种至关重要。
四川大学生命科学学院林辉宏教授及其科研团队于2025年7月14日在著名期刊《Journal of Genetics and Genomics》上发表了名为“A telomere-to-telomere genome assembly of radish (Raphanus sativus L.) provides insights into QTL mapping of bolting traits”的文章,通过对以晚抽薹性状而闻名的白色萝卜品种C60213进行的T2T完整基因组组装,进一步研究与萝卜抽薹相关的QTL定位。

一、高质量基因组组装与评估
作者通过采用长读长测序技术,包括PacBioHiFi(27.91Gb,N50=16.59kb,59×)和ONT(21.25Gb,N50=100kb,44×),成功实现了高质量的基因组组装。混合组装过程由Hifiasm完成,接着使用Purge_dups进行冗余序列清除与杂合性降低。随后通过四轮Racon精修,获得由140条contig构成的初步组装结果,contigN50达到49.69Mb。借助Hi-C数据进一步进行scaffolding,去除较短的contig后保留了135条scaffold,其中13条非冗余scaffold被成功锚定至9条拟染色体上。最终构建出C60213的染色体级别基因组,仅剩4个缺口,并通过手动引入超长ONT数据完成填补,实现了无明显缺口的高完整性组装。
为全面评估基因组组装质量,作者首先采用 BUSCO 对保守植物基因库中的1614个基因进行完整性分析,结果显示99.2%的基因完整保留,表明该基因组具有极高的完整性。随后利用LTR Assembly Index(LAI)评估组装连续性,得分为26.07,显示出较高的连续性水平。此外,通过Merqury工具计算的基因组质量值(QV)显示其错误率低于0.0007%,进一步验证了组装的准确性和可靠性。
基于高质量组装,作者成功注释到了49768个蛋白质编码基因,其中97.38%的基因获得了明确的功能注释,反映出注释的高完整度与准确性。在结构组成方面,约59.72%的基因组序列为重复序列,主要由长末端重复序列(LTRs)构成。LTRs是植物基因组演化中的关键元素,其富集程度与基因组大小密切相关。
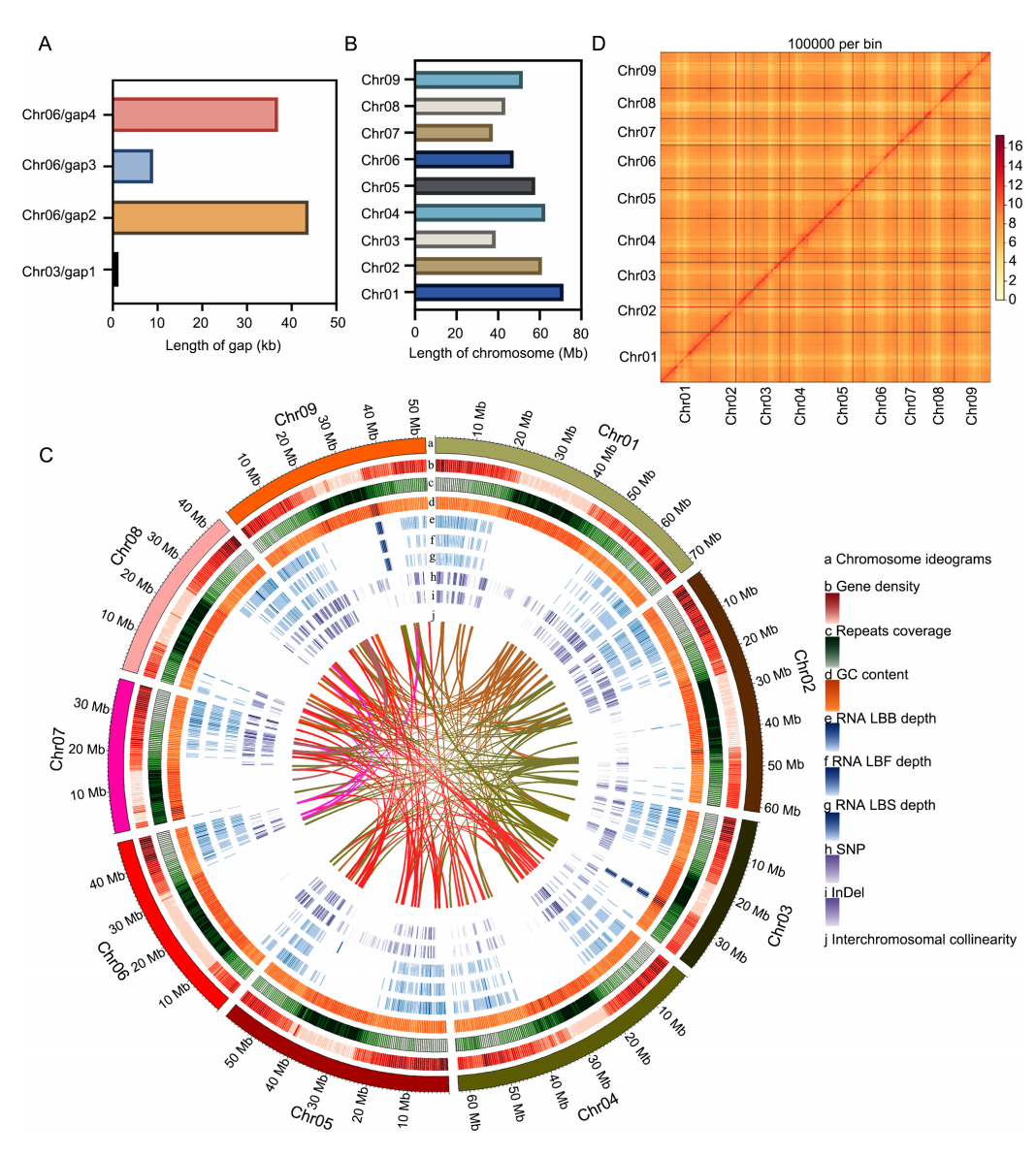
图1:萝卜C60213的基因组测序、组装和评价
二、端粒和着丝粒的鉴定
在高质量基因组组装的基础上,作者成功定位了全部18个端粒和9个无缺口着丝粒。端粒通过识别串联重复序列 TTTAGGG 鉴定,长度从34 bp到1250 bp不等。着丝粒的识别依赖于177 bp 重复序列,它在整个基因组中最为丰富,并经转座元件分析确认具有着丝粒特异性。最终,构建出长度9.38-22.39 Mb的9个CentR序列。进一步分析显示,着丝粒区域富含LTR类逆转座子(如Gypsy、Copia),但基因密度较低,仅包含1567个功能基因,主要参与代谢通路和植物-病原体互作。
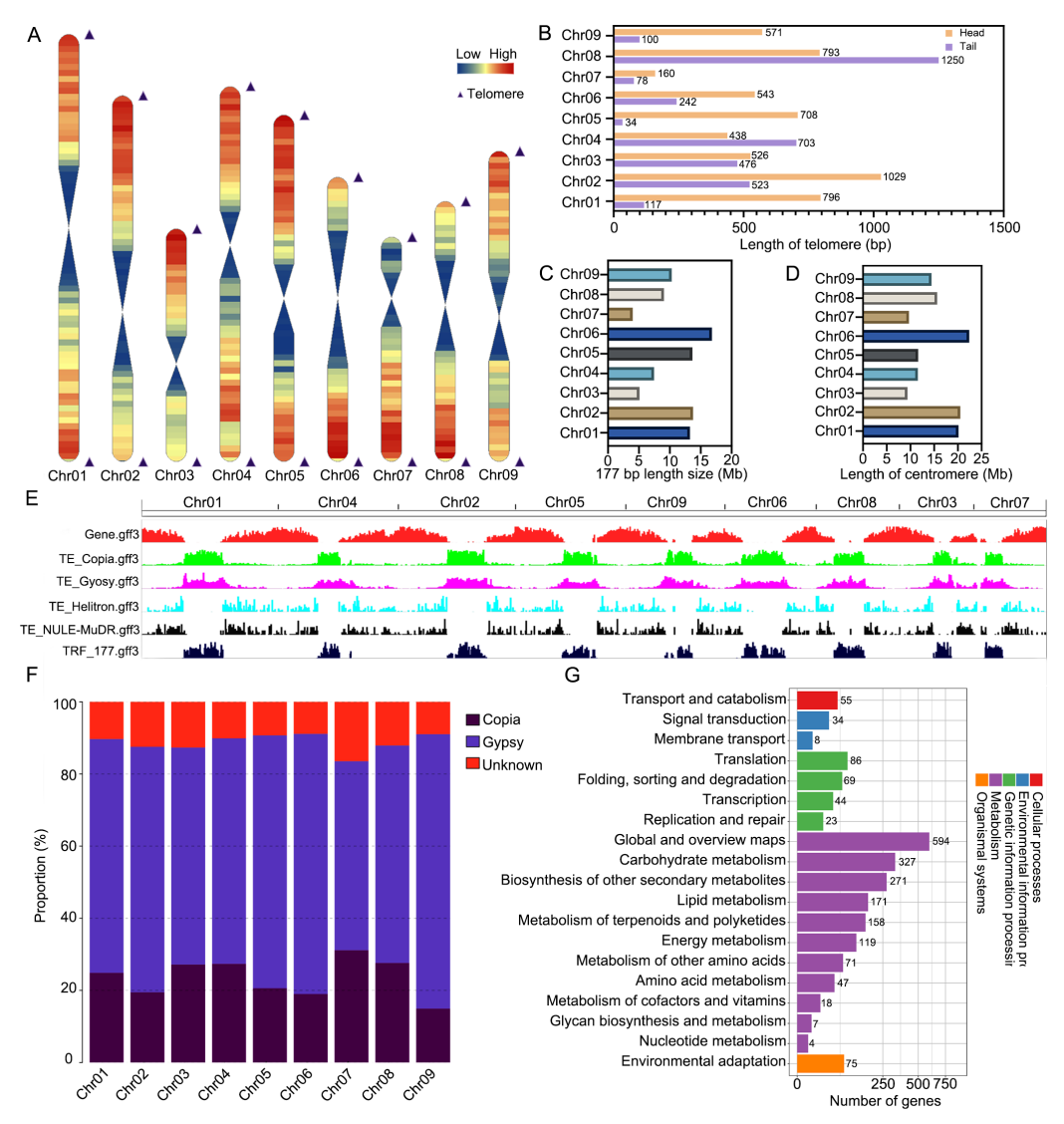
图2:萝卜C60213全基因组中端粒和着丝粒的鉴定和分析
三、全基因组变异鉴定
以C60213基因组为参考,作者进一步鉴定了不同品种萝卜(NAU-LB和RSO5)中的基因组变异,包括单核苷酸多态性(SNPs)、小的插入缺失(InDels)以及结构变异(SVs)。在NAU-LB基因组中,共识别出389521个InDels,其中包含192380个插入和197141个缺失,此外还发现1393613个SNPs。在RSO5基因组中,识别出646879个InDels和2458517个SNPs。
同时,结构变异(SVs)的分析结果显示,少部分SVs位于编码区,这些变异可能会导致基因功能的破坏,并在植物基因组进化中受到强烈的净化选择。此外,GO功能富集分析表明,SVs主要影响萝卜的分子功能和特定代谢通路,这可能是导致不同萝卜品种表型和功能差异的关键因素之一。
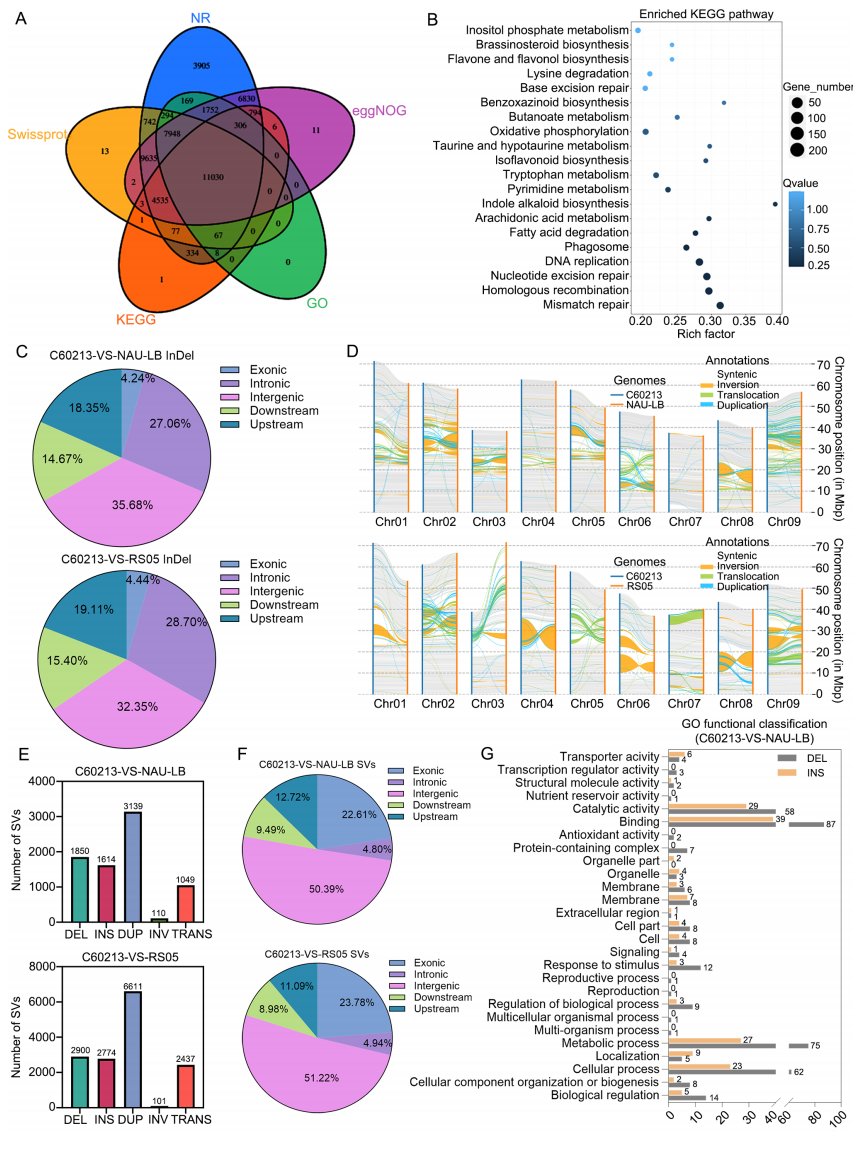
图3:萝卜C60213基因组的基因注释及全基因组变异分析
四、比较基因组分析
在对萝卜基因组的比较分析中,作者发现萝卜与拟南芥、油菜、甘蓝等13个物种具有8972个共同的基因家族,并拥有640个独特的基因家族。通过系统发育分析,萝卜大约在1082万年前与甘蓝和油菜分化,且与黑芥菜的共同祖先较为接近。基因家族进化分析显示,萝卜中有1986个基因家族扩展,4474个基因家族收缩。通过对全基因组复制(WGD)事件的分析,发现萝卜只经历了十字花科物种的三倍化事件。LTR逆转座子插入时间的估算表明,萝卜的LTR逆转座子在约80万年前插入基因组。
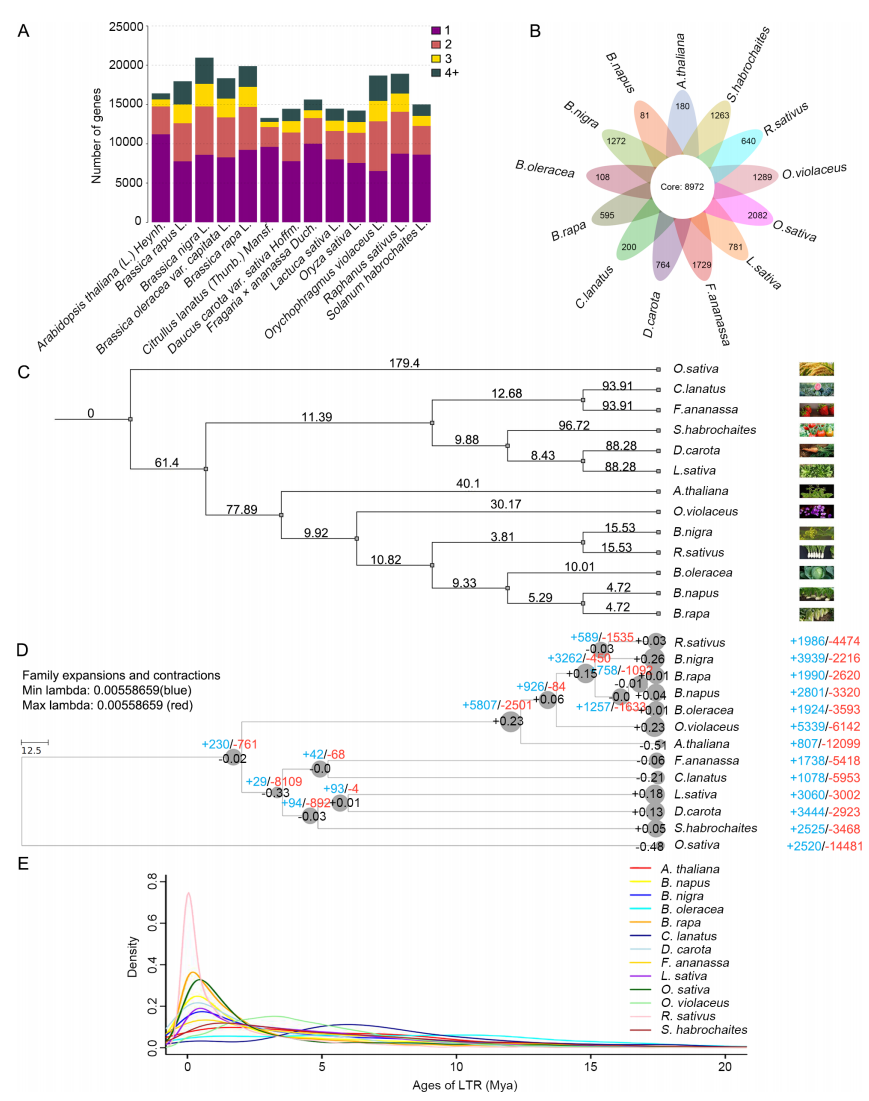
图4:种间比较基因组学与基因组进化分析
五、高密度遗传连锁图谱的构建
对NIL_F2群体的父母和后代进行全基因组测序,成功获得了195.68 Gb的数据,并使用MseI和HaeIII酶组合构建了高质量的文库。C60213基因组总长472.71 Mb,在与亲本对齐后,亲本的平均对齐率超过97%。从4494970个SNP中筛选出了10129个高质量SNP,构建了包含9个连锁群的遗传图谱,总遗传距离为937.41 cM。该图谱提供了详细的标记信息,为后续的基因定位和遗传研究提供了坚实的基础。
六、抽薹和开花相关基因的QTL定位
作者采用来自两个NIL亲本(C60213和C60293)以及217个NIL-F2:3群体的数据,进行了萝卜的抽薹和开花性状的QTL定位。关键性状如抽薹日期(BUD)和开花日期(FD)被选为进一步分析的对象。研究中观察到茎高(FH)、抽薹速度(BS)和抽薹日期(BOD)存在显著的杂种优势效应。利用高密度基因图谱进行QTL定位,识别出与抽薹和开花性状相关的8个主要QTL区间,其中大多数与BUD相关。设置QTL定位的LOD阈值为3,最终在第2号染色体上识别出了这些QTL。
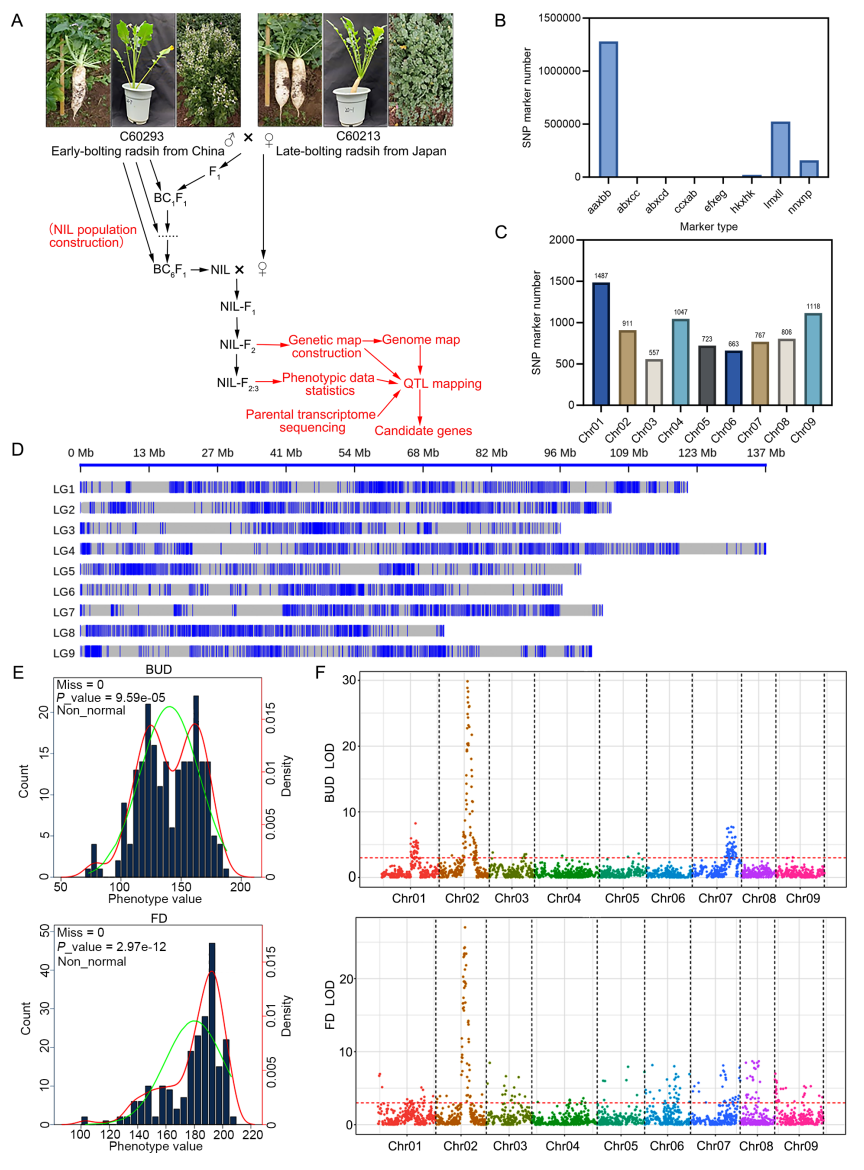
图5:以C60213基因组为参照构建高密度遗传连锁图和数量性状位点定位
七、转录组分析和RsMIPS3的异源过表达延迟抽薹时间
进一步对RsMIPS3基因进行了转录组分析与功能验证,作者发现该基因与抽薹时间密切相关。实验表明,RsMIPS3的过表达显著延迟了抽薹时间,且基因表达水平与RNA-seq分析结果一致。通过与拟南芥基因进行比对,确认RsMIPS3是一个肌醇-1-磷酸合成酶,参与了肌醇的积累过程,从而负向调控抽薹。RT-qPCR分析进一步揭示,RsMIPS3通过调控开花相关基因(如FT、CO、SOC1等)表达,影响萝卜的开花和抽薹时间。
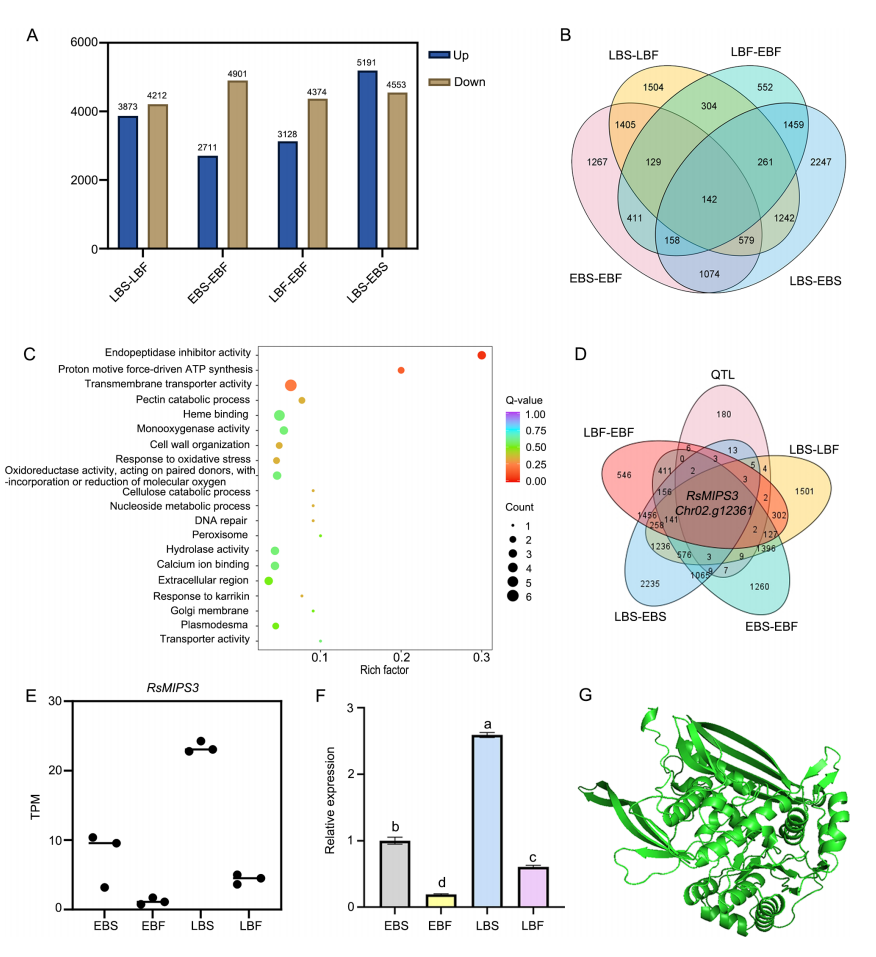
图6:以C60213基因组为参照构建高密度遗传连锁图和数量性状位点定位
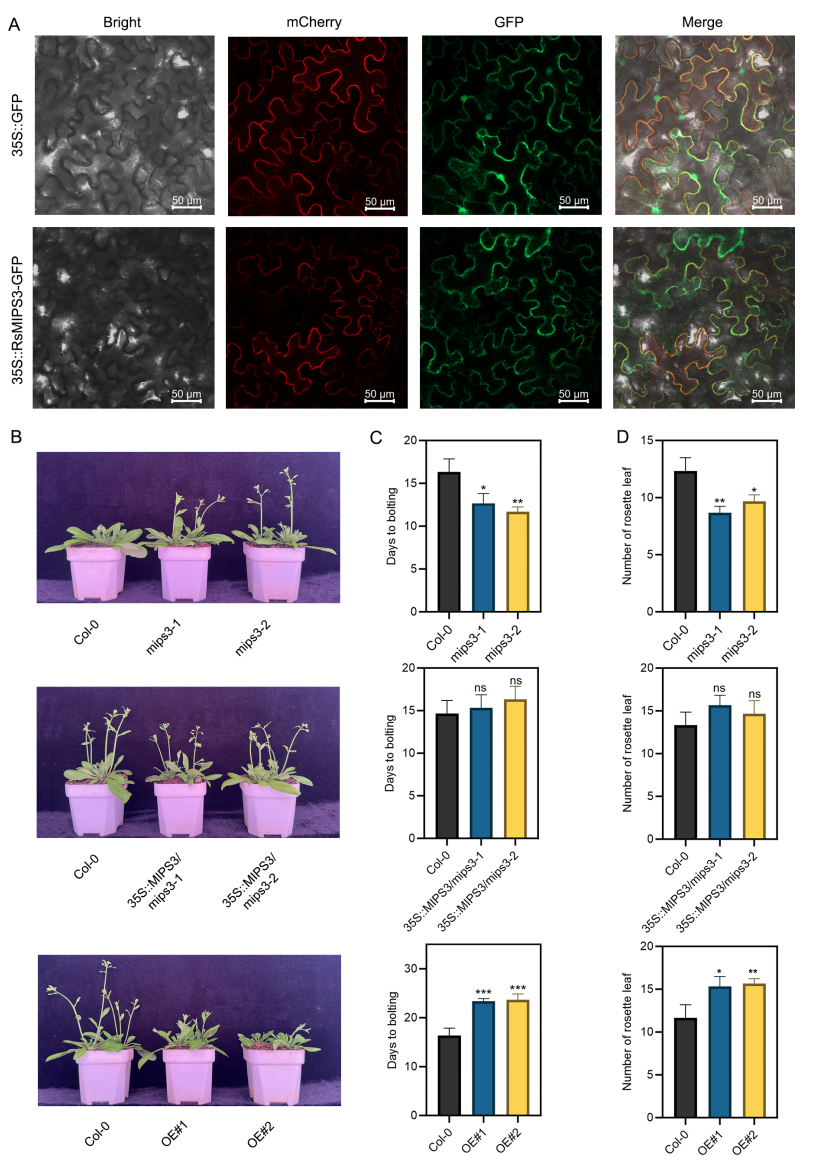
图7:RsMIPS3的亚细胞定位与抽薹调控功能验证
结语
作者对萝卜品种C60213进行了端粒到端粒(T2T)基因组组装,获得了472.71Mb的高质量基因组,注释了49768个蛋白编码基因。通过QTL定位,发现七个与抽薹和开花相关的重要区间,并筛选出关键基因RsMIPS3。该基因在C60213中高表达,且其过表达能显著延迟拟南芥抽薹,验证了其调控功能。研究为萝卜分子育种提供了宝贵资源与理论支持。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!