干货 | 《T2T基因组学系列线上公开课》直播回放&问答汇总
那些曾被称作"基因组暗物质"的迷雾,在Hi-C数据的星轨中逐渐显影。从第一节课的"什么是真正的T2T",到最后一场关于"T2T基因组特色分析"的探讨,我们共同完成了对生命完整性的温柔丈量。《T2T基因组学系列线上公开课》课程终章不是句点而是启动子——您构建的认知质粒正穿梭在学术宇宙,那些被激发的科研噬菌体将持续感染未知领域。
一、问答环节
1.四倍体,基因组接近2G,90条以上染色体,T2T有没有可能
答:如果是异源四倍体,比如油菜和棉花这种,是没有任何问题的,如果是同源四倍体,在加大测序数据量的情况下,我们也是大概率可以组装到四个单倍型的T2T的。
2.杂合度多少算高?
答:一般认为杂合度0.8以上就算较高水平,不过杂合度目前来看,对T2T基因组组装的影响不会像以前那么大。
3.重复序列越多,测序的深度也越大吗?
答:这个需要看情况,如果是正常植物,一般在推荐的测序深度内(50x HIFI+ 50x ONT超长+100x HIC)基本就满足组装T2T需要了,如果是之前项目验证过基因组整体或者部分染色体重复序列非常多和复杂,可以多补充一些ONT超长测序,比如提升到80x左右,我们觉得基本也都足够了。
4.只测HIFI能组装出T2T水平嘛,需要多少数据量
答:目前因为HIFI测序读长的原因,不建议用HIFI组装T2T基因组,如果是因为经费原因考虑,可以尝试下我们最新推出的ONT only的near-T2T方案,我们测试下来效果要比纯HiFi组装基因组好。
5.超长50kb还是100kb会起到决定性影响吗
答:从我们项目经验来看,超长100kb的效果要比50kb的好,如果样本可以满足100kb的建库要求,我们更推荐测100kb,如果是样本没办法达到100kb需要,就再退而求其次选择50kb。
6.请问ONT only的近T2T组装有测试过么?这个跟有HiFi数据的T2T组装的差别会有多大呢?
答:有测试过,小基因组(500M以下)、普通基因组(500M-2G)和大基因组(2G以上)我们都分别测试了,组装完整性要明显好于HiFi的组装结果,但小基因组数据量我们更推荐测到50-80x,大基因组我们更推荐测N50 50kb的数据,数据量也推荐测到50x以上。
7.hic热图呈现的单倍型重复一定是错误的吗?如何判断是否应该保留?
答:一般来说我们需要将hap1+hap2合并挂载HIC,而不是单独挂载HIC,在合并挂载的情况下,我们可以将各自的SV信号“分离”;单倍型之间有SV结合HIC信号和共线性以及Gap的位置即可判断。
8.可以TGS-gapcloser补完,再用quartet补洞嘛?
答:可以的,但是不管用什么软件补Gap,都要明确每个Gap填补的正确与否与对基因组造成的影响(比如,插入太多序列)。
9.两个单倍型之间的存在SV怎么用HIC信号检查呢?
答:先合并挂载HIC,然后结合HIC信号和共线性以及Gap的位置即可判断。
10.rDNA怎么鉴定呢?
答:使用barrnap软件鉴定,这个软件很快速,也比较准。
11.每条染色体的QV是怎么计算得到的?
答:Merqury的中间文件有个.qv文件有每条染色体的。
12.得到最终基因组后的,怎么画的hic热图呀?
答:可以使用haphic的plot模块绘制,这个模块绘制的比EndHIC之类的美观一些。
13.可以用 hic 的数据来做 QV 评估嘛?
答:不可以,最好只用二代评估,此外,最好不要用三代评估QV。
14.着丝粒怎么鉴定?
答:使用Chip-seq数据鉴定是最好的,如果没有的话,可以从串联重复序列以及TE序列的分布来综合判断,目前也有一些软件可以做类似的鉴定,包括TRASH和CentIER。
二、直播回放
扫描下方二维码或在华命生物视频号直播回放中查看。


三、华命科研群
华命生物近期整理了170+篇动植物T2T基因组文献资料,扫码添加工作人员微信,发送单位+名字,管理员验证通过后拉入群聊获取文本全部资料~科研群不仅有动植物的文献包,还有更多科研工具分享,我们会不定期更新文章和科研工具哦!

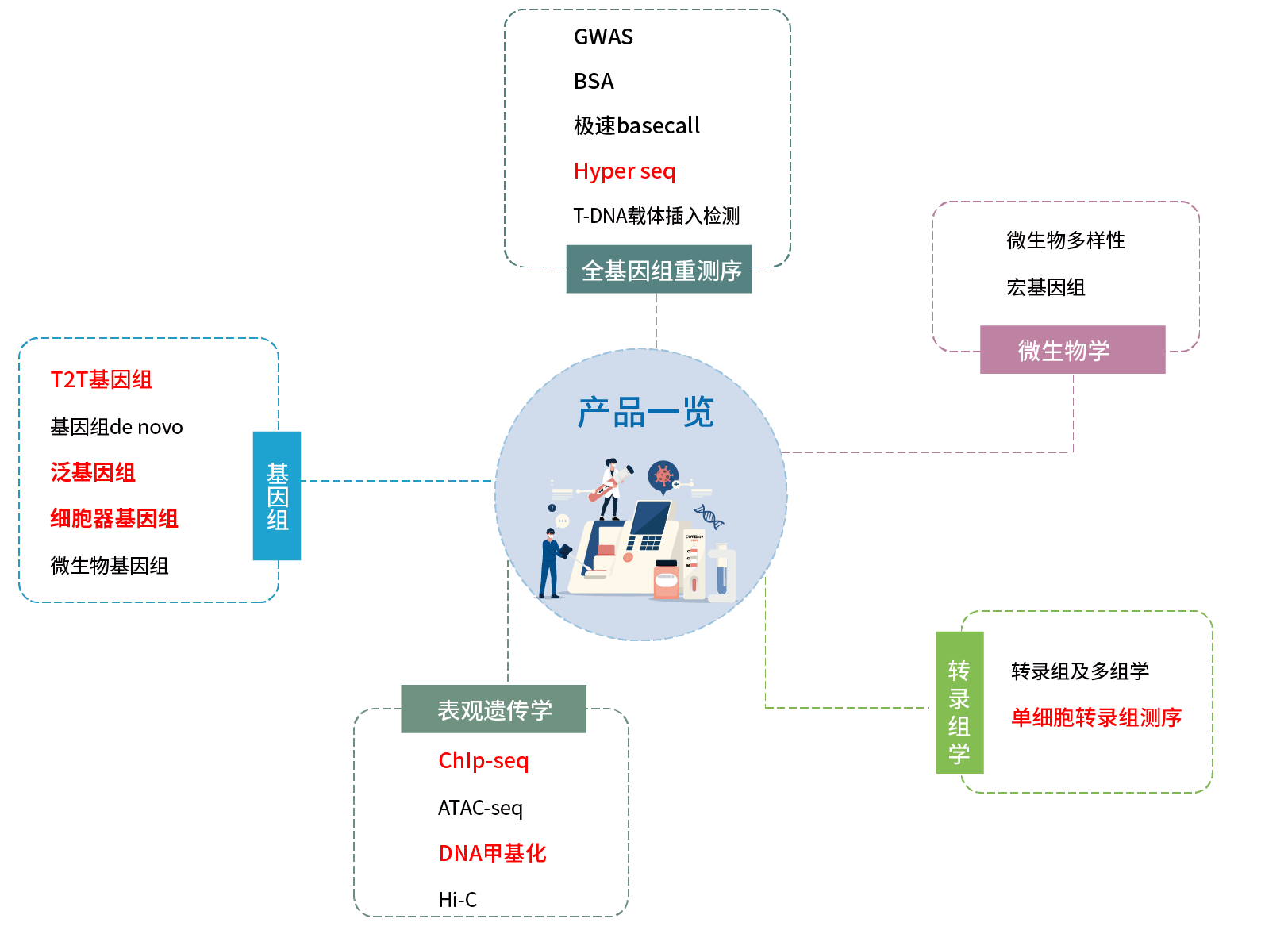
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!