NC重磅 | 莴苣属超级泛基因组发布
栽培莴苣(Lactuca sativa)属于菊科莴苣属,是全球广泛种植和消费的重要蔬菜,富含对人类健康有益的植物营养素。其驯化起源于约4000年前高加索地区的野生刺莴苣(L. serriola)。通过多次驯化和多样化,莴苣发展出了脆叶型、黄油型、罗马莴苣型等多种形态。近年来,基因组研究揭示了莴苣驯化过程中的遗传变异,但结构变异(SVs)及其对驯化性状的贡献仍然不完全明确。
新加坡国立大学沈丽莎研究员及其科研团队于2025年8月6日在著名期刊《Nature Communications》上发表了一篇名为“Lactuca super-pangenome provides insights into lettuce genome evolution and domestication”的重要论文,构建了莴苣属超级泛基因组,揭示转座子扩增与结构变异驱动基因组演化,并解析了关键驯化基因,为莴苣改良提供基因组资源。

一、莴苣代表性类型及其野生近缘种的高质量基因组组装
研究共组装出10种代表性莴苣的基因组,涵盖了五种栽培类型(奶油莴苣、切叶莴苣、长叶莴苣、拉丁型莴苣、油用莴苣)、一个地方品系以及四种野生近缘种。作者采用了PacBio HiFi与Hi-C数据,平均contig N50达到20 Mb,显著优于以往的莴苣参考基因组。组装的基因组大小范围从2.1 Gb到5.5 Gb不等,尤其野生近缘种间基因组大小差异明显。LTR重复序列的完整性(LAI)平均值为19.5,表明重复序列的完整度较好。同时BUSCO完整度达到了96.5%,进一步验证了基因组组装的高质量。通过基因组间共线性分析发现,栽培品种如奶油莴苣的基因组与CrispV11以及其他栽培类型的共线性较高,但与野生近缘种的共线性较低。特别是与 野莴苣L. indica 的共线性差异最为显著,特别在1号、3号及9号染色体上存在大规模的倒位,表现出物种间的遗传多样性。
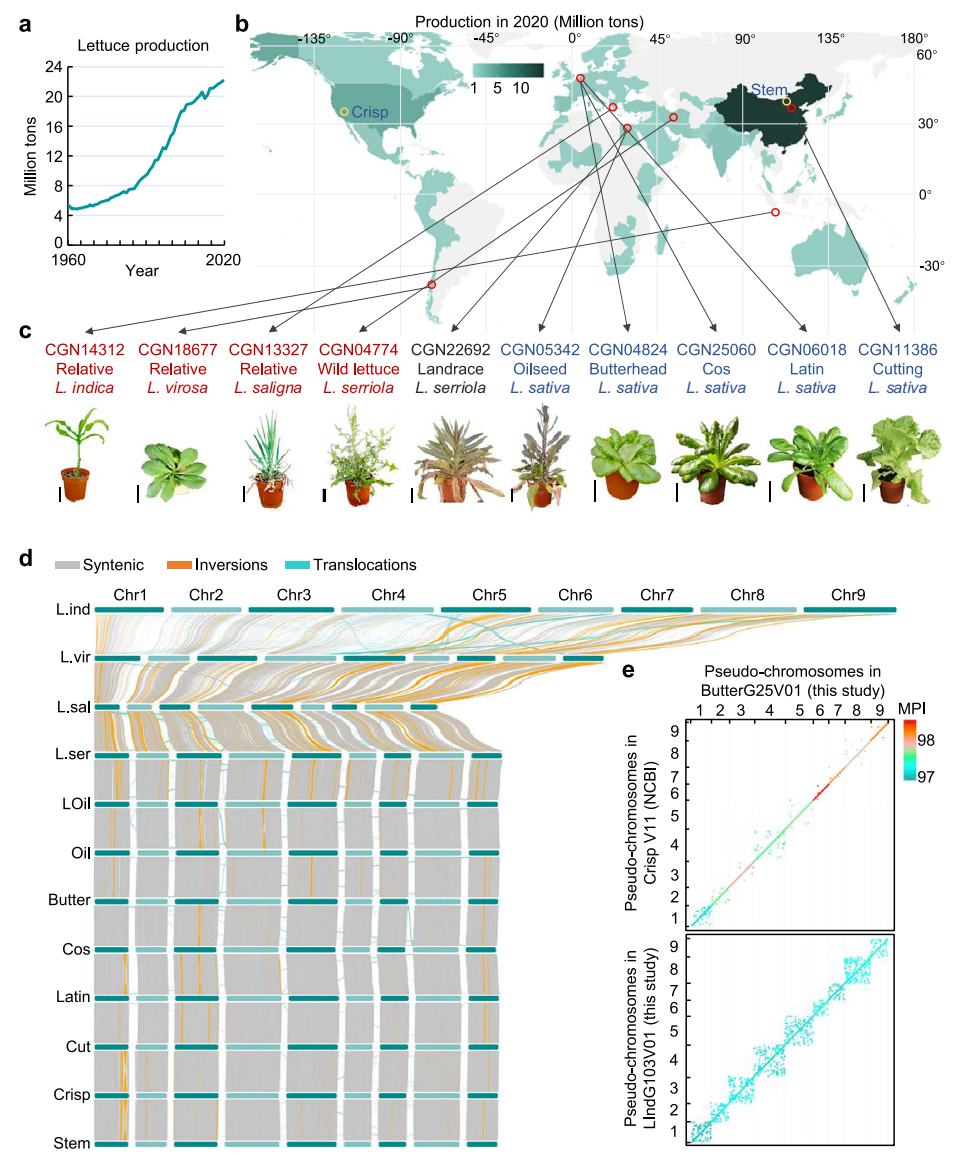
图1:栽培莴苣形态型和野生近缘种的高质量基因组组装
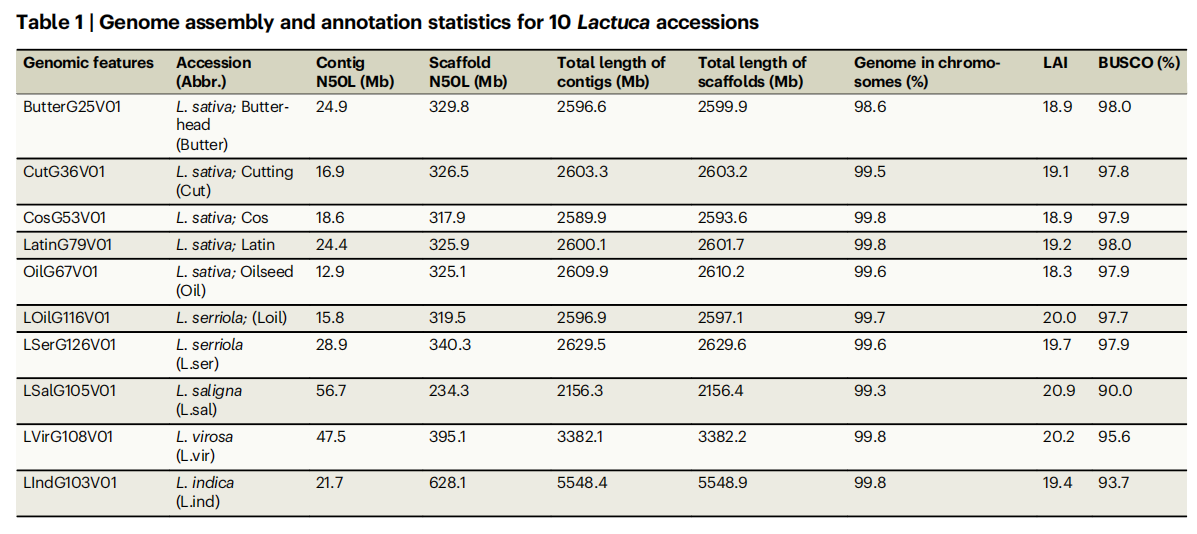
表1:10种莴苣属种质的基因组组装与注释分析
二、莴苣的超级泛基因组
作者整合了12个莴苣基因组以构建莴苣的超级泛基因组,并与已发布的基因组进行了比较。通过聚类分析,定义了37456个泛基因家族,其中42%为核心基因家族,43%为可变基因家族,这表明栽培种与野生种之间存在显著的遗传差异。研究还发现,部分基因家族在栽培种和野生种之间表现出不同的选择压力,驯化保留基因与DNA复制等过程相关,而丢失的基因与温度反应等过程相关。
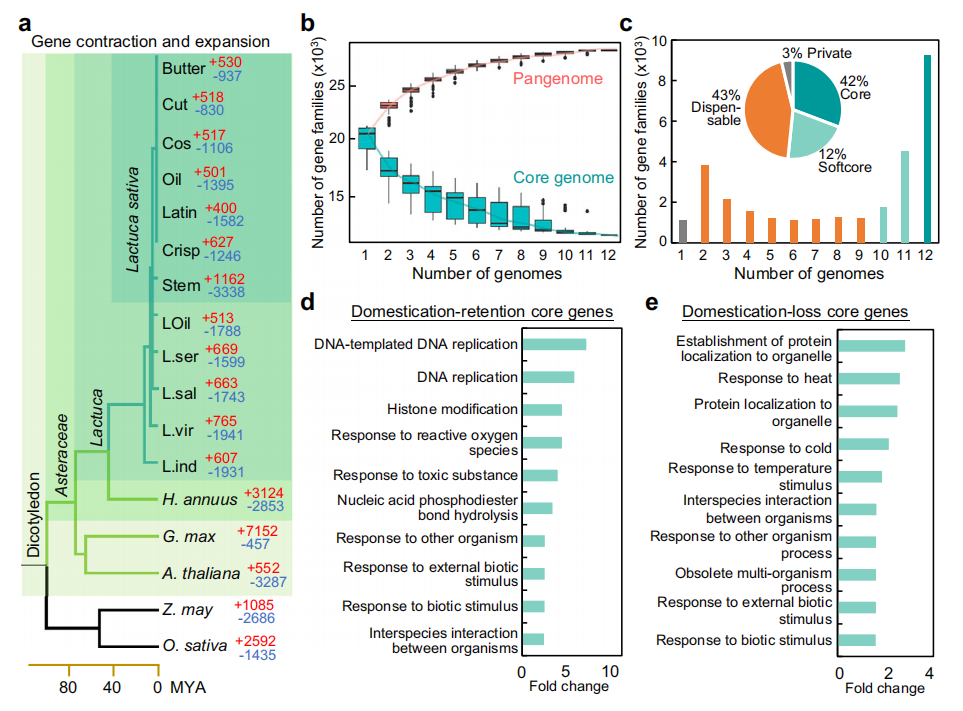
图2:12种莴苣属种质的超泛基因组研究
三、莴苣属中类似的二倍化效应
作者发现,莴苣属基因组虽然较大,但仍表现为二倍体,很可能经历了古倍性化。进一步研究表明莴苣基因组在全基因组翻倍(WGT)后迅速丧失了大量基因副本,类似现象在玉米中也有观察。通过分析重复基因,作者发现约34.8%的基因为单拷贝基因,14.8%为WGT基因,50.4%为小规模重复基因。核心基因中的WGT基因比例明显高于总基因,表明这些基因在莴苣二倍化中可能发挥关键作用。GO分析显示,核心WGT基因参与芽体系统发育和RNA生物合成,且其表达水平显著高于其他重复基因。此外,WGT基因周围CG甲基化水平较低,尤其在转录起始位点附近,暗示低CG甲基化可能有助于这些基因的保留。
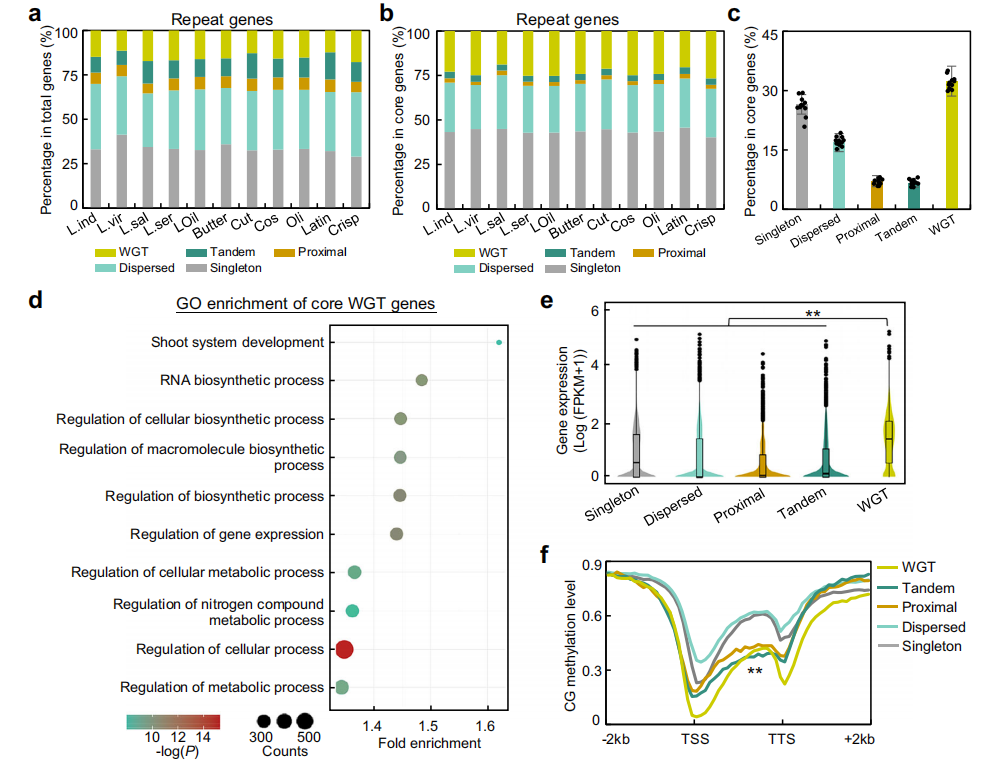
图3:莴苣属中保留的WGT基因的特征
四、转座子扩增有助于基因组大小
作者研究发现,莴苣基因组中约84.5%-89.1%是重复元素,主要为长末端重复(LTR)反转录转座子,其中Gypsy和Copia占大多数,约占总重复序列的82.6%。少部分重复元素为DNA转座子约占2.1%。长叶野莴苣L. saligna的基因组重复序列比例为84.5%,为最小,而野莴苣基因组的比例最高,达到89.1%。进一步研究发现,野莴苣的完整LTR比例为28.2%,明显高于长叶野莴苣的17.9%。大部分完整LTR长度约为8.0 kb,但野莴苣的LTR较短,且具有较多6.3 kb的完整LTR。尤其是Copia和Gypsy的LTR在野莴苣中的插入时间显著较年轻,表明近期的LTR扩展,经过多轮插入,促成了野莴苣基因组的显著膨胀。此外,野莴苣基因组中的CHH甲基化水平显著低于黄油头莴苣,尤其在LTR区域。通过对CMT2同源基因的研究,发现野莴苣中CMT2表达水平较低,进一步表明CMT2的转录水平可能与LTR扩展及基因组大小的增加密切相关。
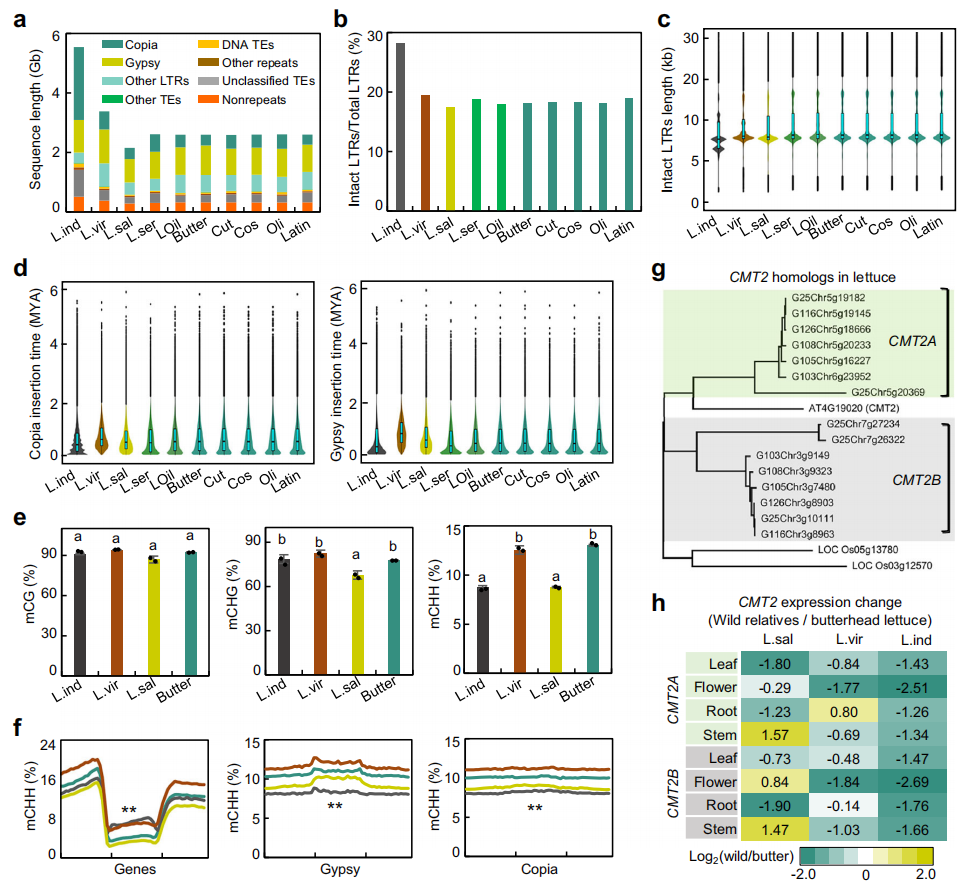
图4:转座子扩增对基因组大小的贡献
五、转座子驱动的结构变异
作者通过对12个莴苣基因组的比对,识别了大量结构变异(SVs)。在栽培莴苣中,切叶莴苣、菜心莴苣、拉丁莴苣和脆叶莴苣的基因组平均识别出87,625个SVs,而油菜莴苣和茎叶莴苣的数量更高,分别为123841和192,230个。野生亲缘种中,L. serriola、L. indica、L. virosa和L. saligna分别识别出131682、112055、244996和294989个SVs。大部分识别的SV为缺失/存在变异(PAVs),其中42.0%为插入,50.3%为缺失。PAVs主要分布在重复区域的边界,尤其是LTR丰富的异染色质区,表明SV事件可能与转座子活动相关。PAVs与基因表达水平相关,带有PAVs的基因表达水平普遍低于没有PAVs的基因。比如,L. indica中的CMT2A基因含有大量PAVs,其表达水平较低,符合其CHH甲基化水平下降的趋势。
此外,RLL2A基因的PAV在红叶菜心莴苣和拉丁莴苣中存在,而在其他莴苣类型中缺失。研究表明,RLL2A的过表达可提高叶片花青素含量,表明PAVs可能导致莴苣叶片花青素含量的变异。这些发现揭示了转座子扩展和PAVs在莴苣基因组变异中的重要作用。
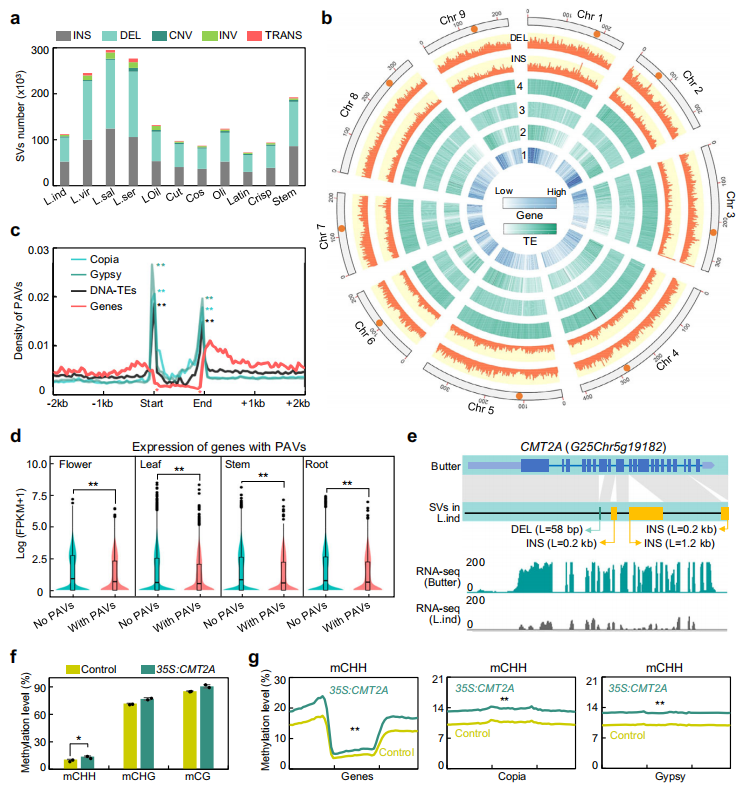
图5:转座子驱动的结构变异
六、结构变异与莴苣驯化的关联
作者发现,栽培莴苣在驯化过程中积累了大量与结构变异相关的PAVs,主要分布于基因间区,而仅约6.8%位于基因区域。研究共鉴定出506004个驯化相关PAV簇,包括核心型(20.5%)、可变型(52.8%)和特有型(26.7%),显示不同栽培型共享一组核心PAVs,符合其单一祖先起源。作者进一步鉴定出3232个核心相关基因,这些基因富集于春化反应、胞内运输及细胞壁合成等通路。尤其是开花抑制因子FLC在栽培叶用莴苣中拷贝数扩增至5-8个,而野生型仅有3个,与栽培型莴苣普遍晚花的性状一致。新增FLC拷贝在野生基因组中多表现为大片插入或缺失,提示SV驱动基因拷贝变异并在驯化过程中被选择,以延迟开花、延长采收期并提高产量。此外,作者还发现与细胞分裂及胁迫响应相关的基因变异,包括一个超过90 kb的缺失区。
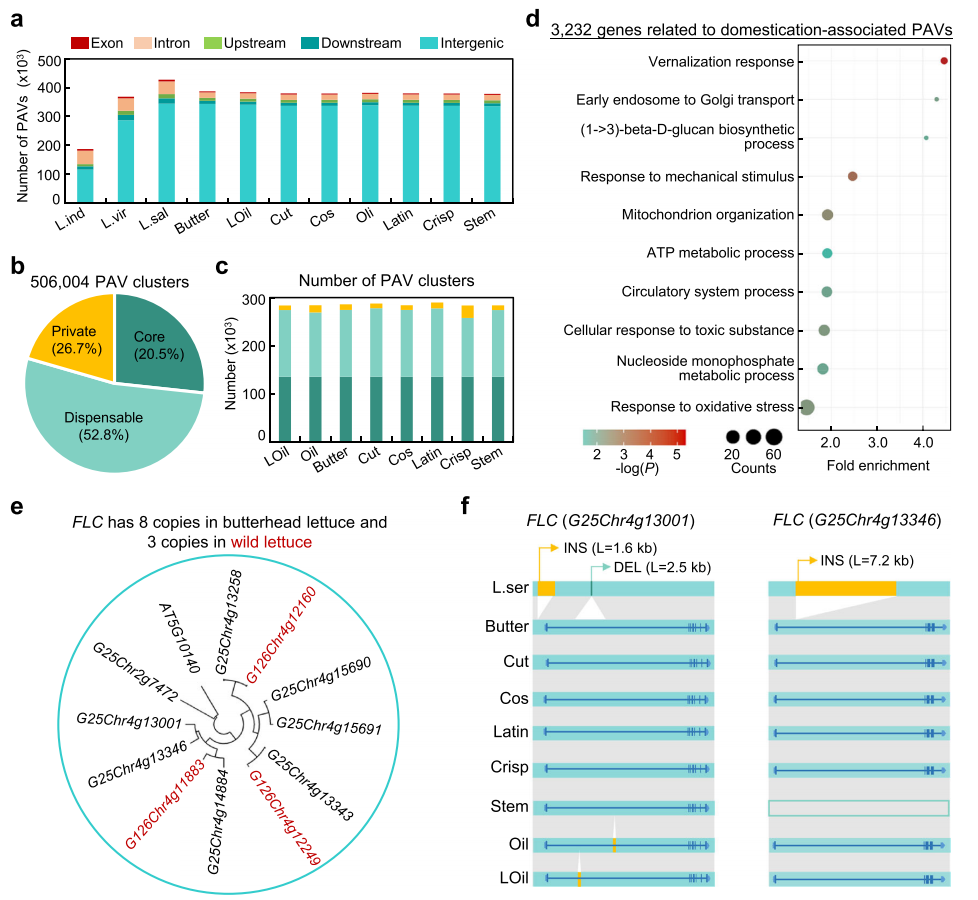
图6:与莴苣驯化相关的PAV鉴定
结语
本研究以12个莴苣属物种的染色体级基因组为基础,构建了高质量的超级泛基因组和图谱化参考。结果显示,莴苣基因组大小差异极大,主要由转座子尤其是LTR逆转录子扩增驱动。其中野莴苣近期发生多轮LTR爆发,并伴随CHH甲基化水平下降与CMT2表达削弱,揭示了基因组膨胀的分子机制。通过图谱参考解析发现,结构变异尤其是缺失/存在变异(PAV)广泛分布于转座子与基因交界区,并可抑制邻近基因的表达,反映了变异与基因调控间的复杂关系。在驯化过程中,核心基因集趋于保守,但部分与春化和开花调控相关的基因如FLC发生拷贝数扩增及插入缺失,赋予栽培莴苣普遍晚花、延长采收期的特征。这一研究不仅深入揭示了莴苣属的演化动力,也为目标性状的功能基因解析和分子育种奠定了坚实基础。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!