Plant Commun重磅 | 首个茄子无间隙T2T基因组
茄子 (Solanum melongena L.) 隶属于Solanum属(茄科),在全球茄科作物的产量中排名第三。尽管到2024年为止已经发表了四个栽培茄子基因组和两个野生茄子基因组,但仍存在如端粒和着丝粒这样大量缺口和未组装的复杂区域。填补这些空白,为基因挖掘和功能研究、标记辅助育种以及进化生物学提供更多连续完整的茄子基因组序列至关重要。
浙江省农科院蔬菜研究所包崇来研究团队在国际著名期刊Plant Communications上发表名为“A complete telomere-to-telomere genome assembly of Solanum melongena uncovers key regulators in pan-tissue anthocyanin biosynthesis”的研究论文,报告了第一个无间隙的茄子T2T基因组组装,并分析了关键调控基因家族SmeMYB的跨组织表达特征。

主要研究内容
在本研究中,作者采用多平台策略,完成了茄子自交系‘HQ-1315’的无缺口端粒到端粒(T2T)基因组组装。作者首先利用PacBio HiFi(51.36 Gb)和 ONT(51.10 Gb)数据进行初步组装,再结合Hi-C数据(131.73 Gb)将 scaffold 锚定到12条染色体上。随后,作者通过ONT数据比对,并参考最新的 Smel NO211 基因组,成功补齐40个缺口,实现T2T水平的组装。最终,作者获得了12条伪染色体,总长1161.12 Mb,contig N50达到53.47 Mb。BUSCO分析显示完整性为 99.50%,一致性质量值(QV)为 43.67(对应99.99% 的准确度),表明该基因组具有极高的完整性与精确性。
作者基于oligo-FISH 技术构建了茄子基因组的细胞遗传学与物理图谱。在12 条染色体的16个区域设计了2.4万个探针,并成功实现染色体核型整合。基因分布分析显示,基因在染色体末端区域更为密集,端粒重复序列与rDNA位点均得到验证。与早期组装版本相比,作者构建的Smel HQ v2.0修正了1、10、11号染色体的倒位,并填补了4号染色体的缺口,显著提升了组装精度。进一步比较了 Smel HQ v1.0、Smel 67/3 v4.1 和 Smel HQ v2.0三个版本,发现它们在染色体结构上存在差异。通过设计特异性探针库并进行FISH验证,证实Smel HQ v2.0成功修正了三个倒位并填补了一处缺口,从而大幅提高了基因组组装质量与准确性。
在 Smel HQ v2.0 基因组中,作者共预测到35004个编码基因(97.66%获注释),基于559个单拷贝基因的系统发育分析,作者推测茄子与非洲茄(S.aethiopicum)约在770万年前分化。在茄属进化过程中,共有61个基因家族发生扩张,275个收缩。作者进一步分析了MYB转录因子家族,鉴定出213个成员(104个R2R3-MYB、104个1R-MYB和5个3R-MYB)。在不同颜色茄子组织的转录组数据中,作者发现分别有142、142、138、58和16个SmeMYB在茎、花、萼片、果皮和叶中差异表达,其中8个在所有组织中均有表达。值得注意的是,SmeMYB182在深紫色组织中高表达,而SmeMYB175特异性地在深紫色萼片中高表达。部分SmeMYBs(如SmeMYB165和SmeMYB91)在特定组织中与花青素积累密切相关;在10个与花青素合成相关的SmeMYBs中,作者发现6个主要在花中高表达,3个在叶中高表达,SmeMYB33则在多个组织中广泛表达。这些结果为解析茄子花青素积累机制及分子育种提供了重要依据。
此外,作者构建了首个整合7个茄子基因组(5个栽培种和2个野生种)的开放数据库(http://47.92.172.28:12068/Eggplant/home/index),提供基因检索、序列比对与共线性分析等功能,为茄子研究提供了一站式信息平台。
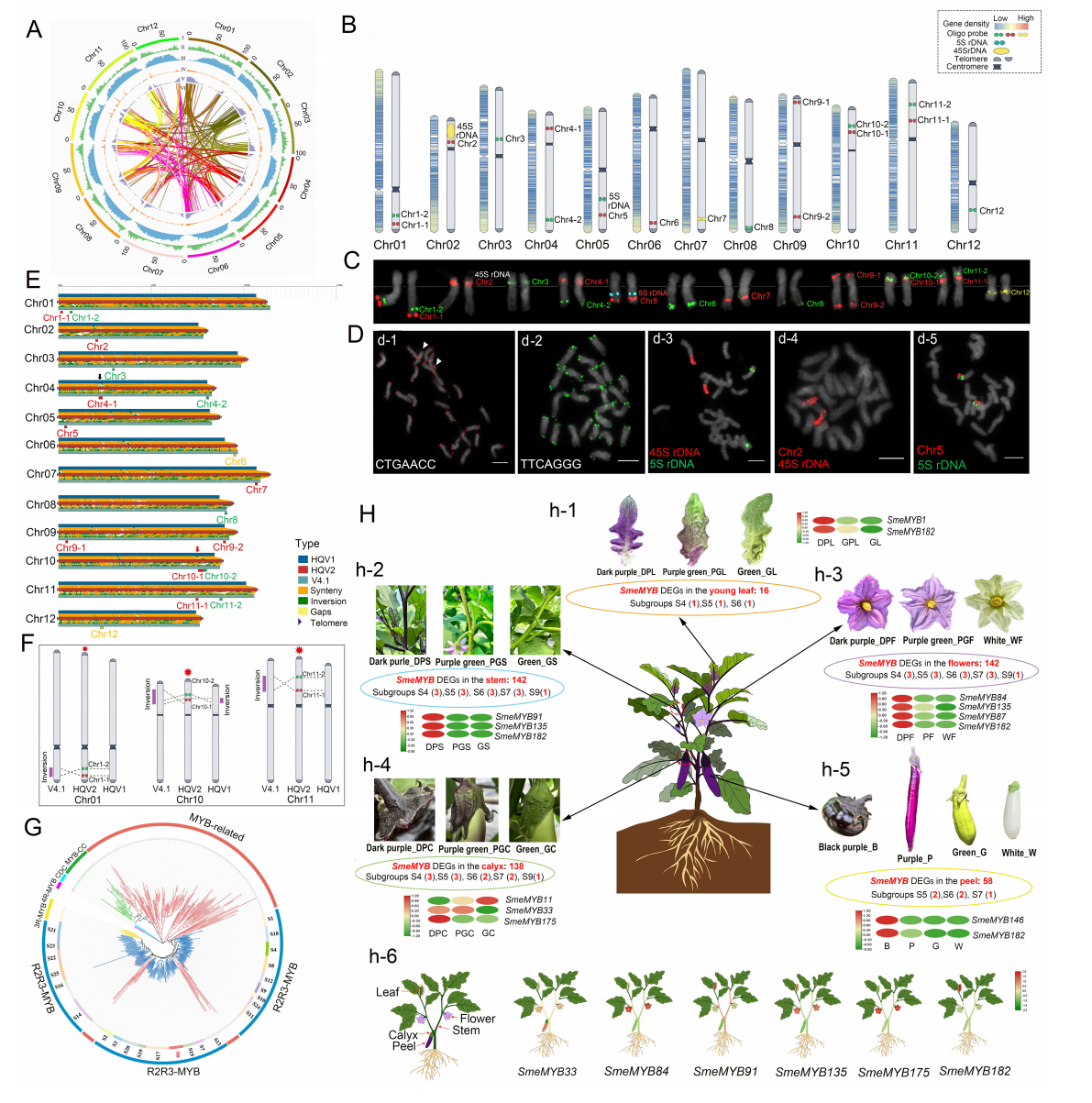
图1:Smel HQ v2.0 基因组的段落到段落组装
结语
本文构建了一个高质量、无间隙的茄子T2T基因组(Smel HQ v2.0),并通过细胞遗传学方法验证、纠正染色体结构问题。基于此基因组,作者鉴定并分析了关键调控基因家族SmeMYB的跨组织表达特征,还搭建了公开的茄子基因组数据库,便于科研社区访问与应用。这项工作为茄子基因功能研究、育种和表型解析提供了更精确、完整的基因组基础,推动深入分子机制研究与应用转化。

