NC重磅|牛T2T基因组解析X染色体新着丝粒形成
近年来,牛基因组研究不断推进,但由于包含大量重复序列,尤其是性染色体,其完整端粒到端粒(T2T)组装长期面临技术挑战。现有超过20个牛基因组版本均未包含完整的X染色体,这限制了对其在智力、精神功能、生殖及产奶量等关键性状中的作用进行深入研究。同时,传统参考基因组存在缺口,无法代表牛的全部遗传多样性,并造成结构变异识别的偏倚。
澳大利亚阿德莱德大学的研究团队于2025年11月28日在国际著名期刊Nature communication上发表了一篇题为“Insights into natural neocentromere evolution from a cattle T2T X chromosome”的研究论文,首次构建了来自和牛(Wagyu)的无缺口T2T X染色体及四条完整常染色体,并系统解析其着丝粒结构,发现其可能为天然形成的进化性新着丝粒,为理解着丝粒演化提供重要线索。

一、基因组组装与注释
研究整合 PacBio HiFi(58.1×)、ONT 普通长读长(121×)、ONT 超长读长(18.3×)、HERRO 校正 ONT(57×)、Hi-C及 Illumina 短读长(F1: 81.8×、双亲: 78×)等多平台深度数据,构建了高质量的单倍型解析基因组。通过多种组装策略比较与严格筛选,最终获得长度 3.14 Gb、仅含 17 个缺口、BUSCO 完整度达 99% 的优质组装,并成功生成五条端粒到端粒(T2T)染色体,其中包括一条完整的牛X染色体。
新组装较现有参考基因组ARS-UCD2.0增加了431Mb的序列,显著提升基因组的完整性与连续性。分析显示,常染色体残余不完整主要源于着丝粒及rDNA区域的复杂重复导致的组装图缠结。此外,未定位序列中约86.8%为重复序列。
UOA_Wagyu_1基因组包含51%的重复序列,较ARS-UCD2.0的41%明显增加。在和牛中共检测到9个完整rDNA拷贝及190个未定位拷贝,复杂的rDNA阵列也阻碍了BTA4的完全组装。牛的rDNA单元约35kb,由高度一致的18S、5.8S、28S及变异显著的IGS组成。
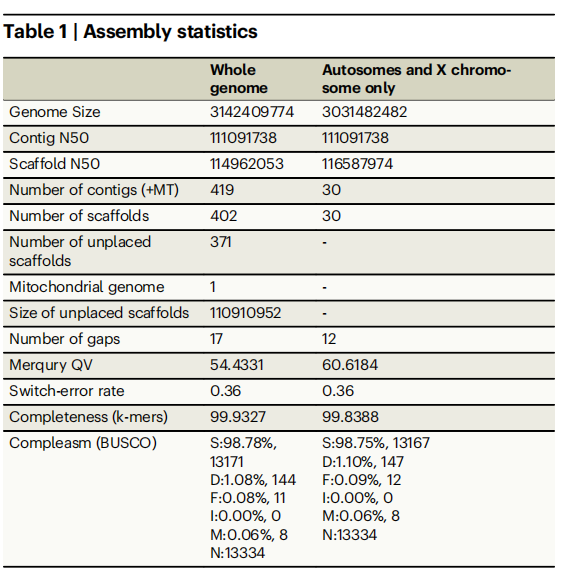
表1:装配统计数据
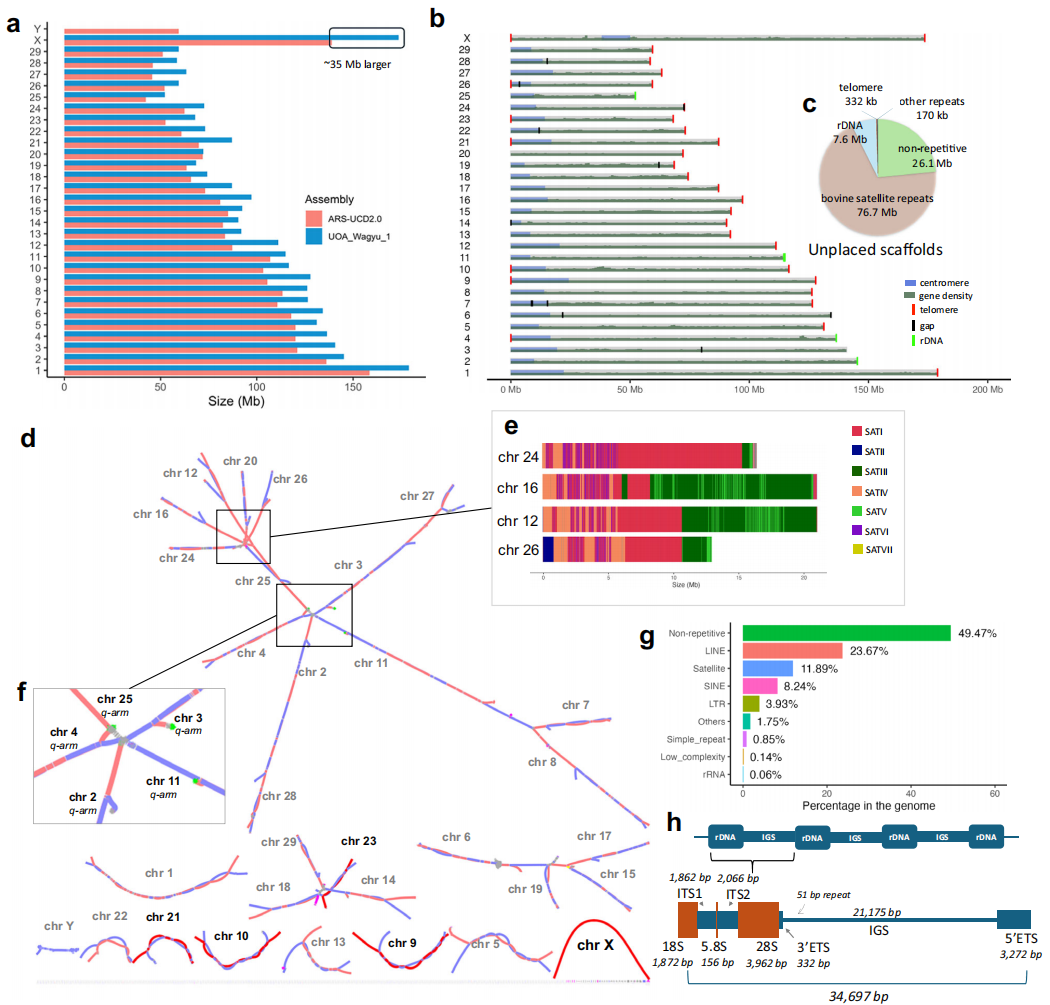
图1:UOA_Wagyu_1基因组组装图
二、染色体的p臂和q臂上的端粒
由于目前尚无牛基因组在染色体两端均实现完整端粒解析,且多数组装缺乏对端粒重复序列的系统分析,作者对和牛基因组的端粒完整性进行了详细评估。结果显示,该基因组包含5条T2T染色体、1条T gap T染色体、19条仅一端具有端粒的染色体,以及5条两端均缺乏端粒的染色体。整体上,和牛基因组共检测到64,965个端粒单元,远高于参考基因组ARS-UCD2.0的6,849个。此外,有31条未定位的scaffold含端粒重复,总长度达26Mb。组装染色体的平均端粒长度为6.8kb,而PacBio HiFi原始测序数据中的平均端粒长度为3.1kb,表明组装显著提升了端粒区域的完整性。
三、着丝粒卫星重复序列的基因组结构
牛着丝粒区域由多种卫星重复序列构成,由于近端着丝粒染色体含大量长串联重复,特别是BTA12、BTA16、BTA24与BTA26的结构高度相似,导致这些区域在组装中形成复杂缠结,难以跨越着丝粒至端粒。尽管如此,本研究成功获得五条跨越着丝粒的常染色体组装。分析显示,约84%的着丝粒由七类卫星重复组成,其中SATIII具有最强的CENP-A富集信号,提示其可能具备核心着丝粒功能。不同染色体间的卫星重复分布与拷贝数存在差异,但已解析的所有常染色体着丝粒均呈现统一的排列顺序:端粒、SATVII(伴随SINE/LINE和简单重复)、SATII、SATIV。根据SATI与SATVI的夹杂模式,常染色体着丝粒可分为两类结构类型,为理解牛着丝粒组织和演化提供重要依据。
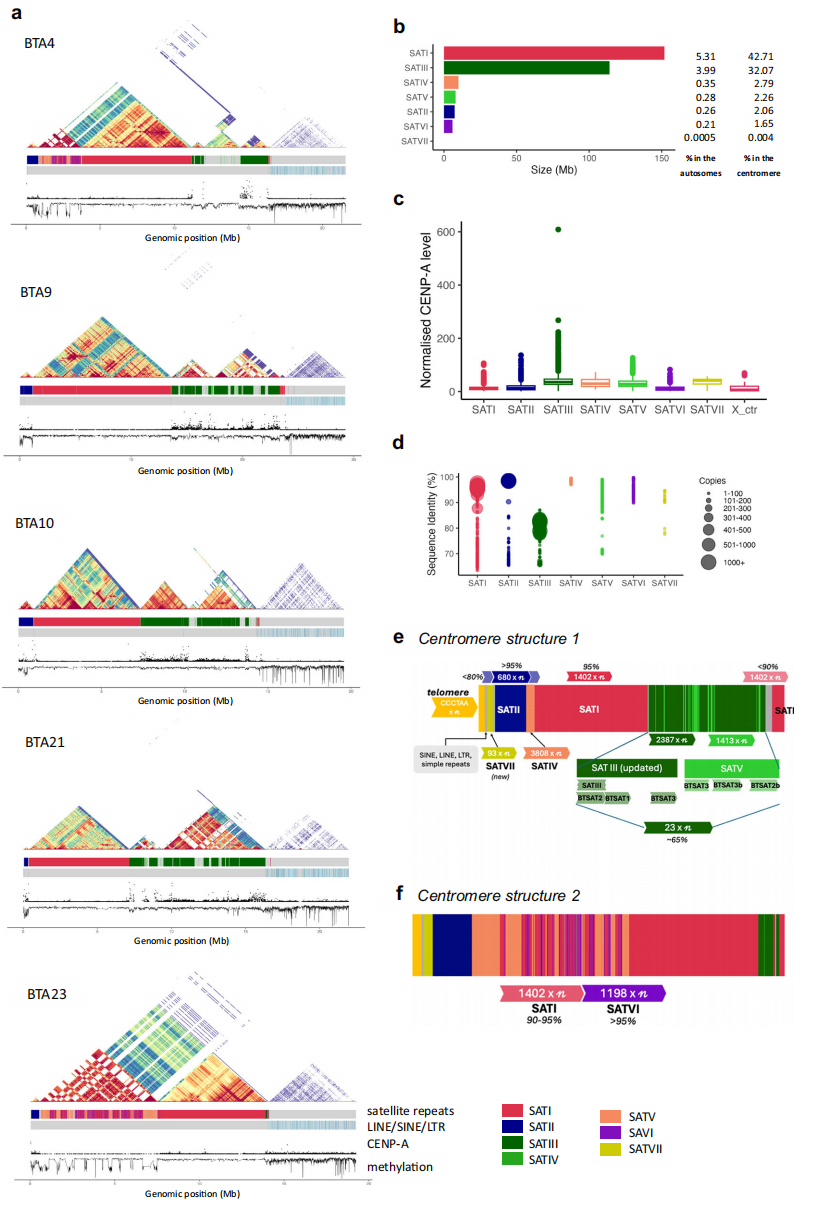
图2:UOA_Wagyu_1 常染色体着丝粒及卫星重复序列概览
四、BTAX 着丝粒中的倒置重复与性别特异重复
本研究将BTAX着丝粒精确定位于38-50Mb区间,并揭示其结构与常染色体着丝粒显著不同。该约12Mb的区域中88.7%由高度一致的倒位重复构成,其中72.2%为转座元件,重复长度范围从10bp延伸至620kb。与典型牛着丝粒富含卫星重复的特征不同,BTAX着丝粒几乎缺乏卫星DNA,且CENP-A富集水平低,支持其作为天然形成的新生着丝粒的观点。
作者进一步解析该区域的重复块结构,鉴定出两类小重复(2,898bp与6,558bp,共217拷贝)以及九段318kb的超长重复,二者合计覆盖约24%。此外,研究还识别出两个BTAX特异性重复(XCTR4、XCTR5),并确认Bisbis-1.8为该着丝粒中最丰富的转座元件。
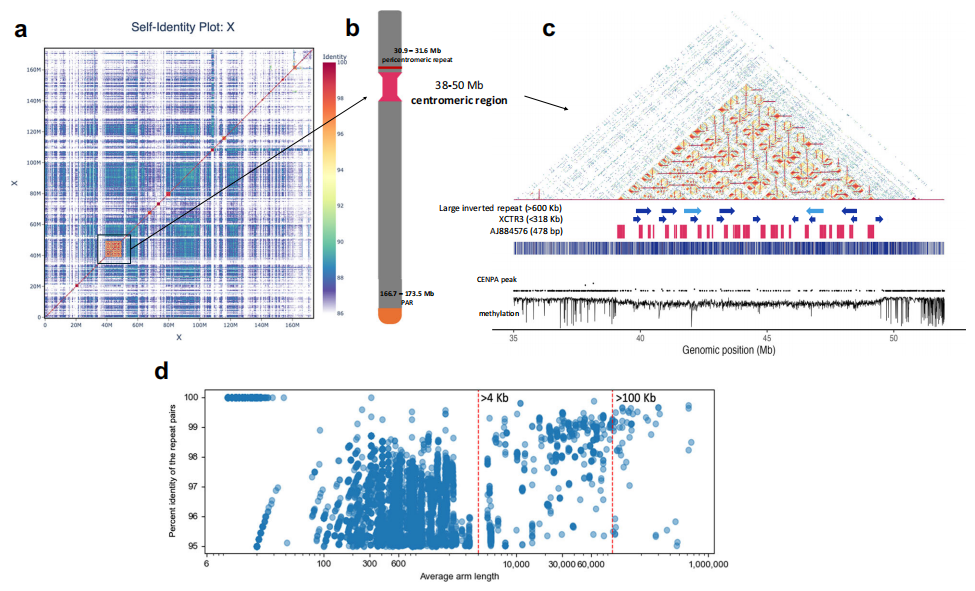
图3:牛X染色体结构
五、BTAX 着丝粒呈现独特的 CpG 特征
本研究揭示了 BTAX 着丝粒独特的 CpG 甲基化特征,其表观遗传模式明显不同于典型着丝粒。基于 UOA_Wagyu_1_Y 基因组的甲基化分析显示,BTAX 着丝粒的 CpG 甲基化水平显著低于非着丝粒区域,差异约 10%,且该区域 CpG 含量远低于预期,CpG 与非 CpG 二核苷酸的比例亦显著低于常染色体着丝粒,而 TpG 的高频出现 进一步表明 CpG 在此区域已发生大幅度减少。相比之下,人类着丝粒中实际 CpG 数量与理论预期呈较强相关,而牛 BTAX 着丝粒则偏离这一模式,说明物种间着丝粒 CpG 保留与去除机制存在显著差异。
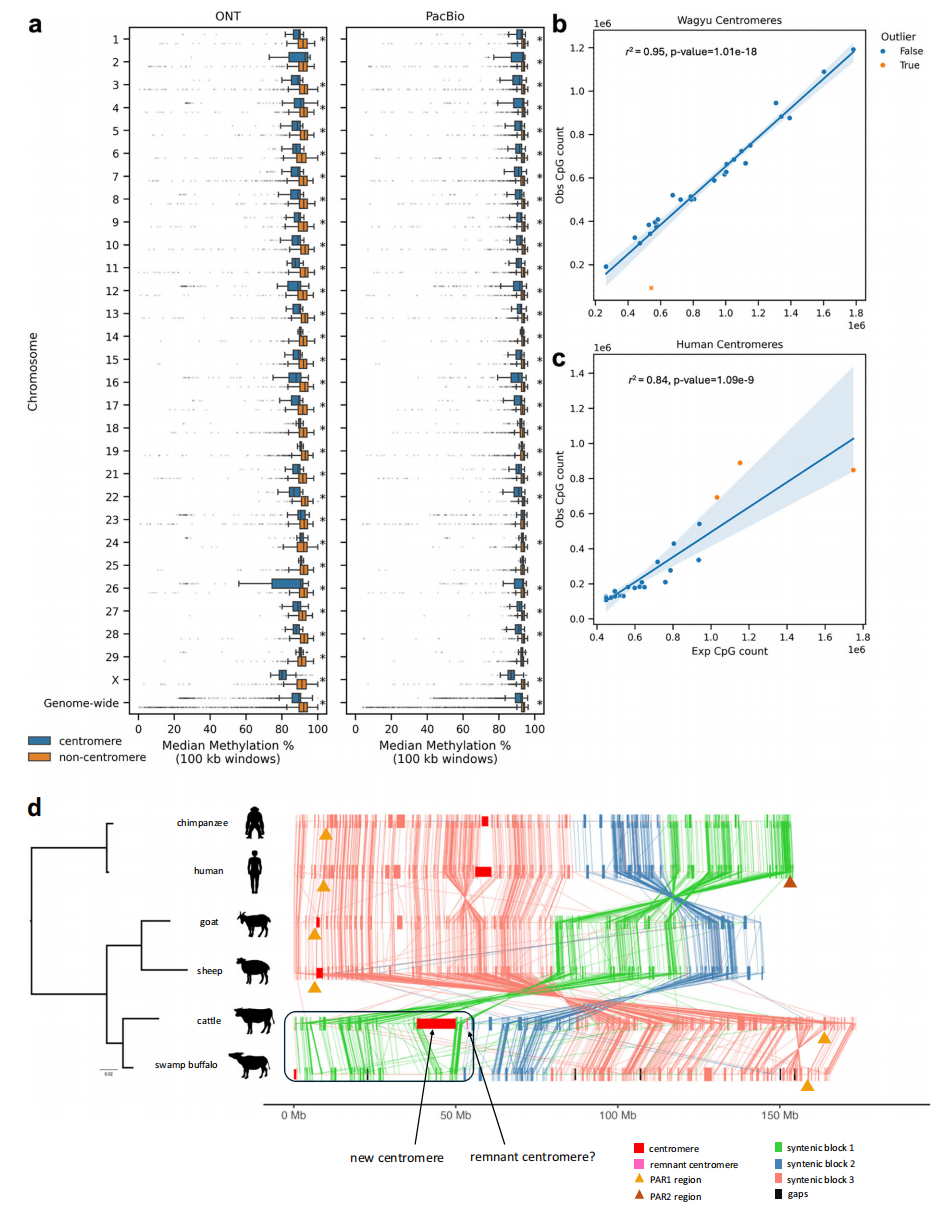
图4:着丝粒和CpG甲基化
六、X着丝粒的动态进化
种间比较显示,X染色体着丝粒在灵长类与牛科间发生动态演化。人类、黑猩猩、牛、水牛、绵羊和山羊中可见四种着丝粒位置。绵羊与山羊共享祖先位置,而牛与水牛均发生偏移。水牛着丝粒仍含0.8Mb牛科卫星序列,而牛(BTAX)为次中部着丝粒,并在距着丝粒3.5Mb处保留SATII残迹,可能反映祖先着丝粒的迁移历史。
七、牛X-YPAR与非PAR共线性
牛的X-Y假常染色体区(PAR)具有显著的物种特异性。X-PAR位于Xq末端,Y-PAR位于Yp,并为多个比较物种中最长。两者包含31对同源基因,另有牛特有的PRP基因位于Y-PAR。牛在PAR中拥有灵长类缺乏的BDA20与OBP,并缺失多种灵长类共有基因。Y-PAR向外包含X-退化与扩增区,分别保留与X的同源基因或仅存在于Y的扩增基因。
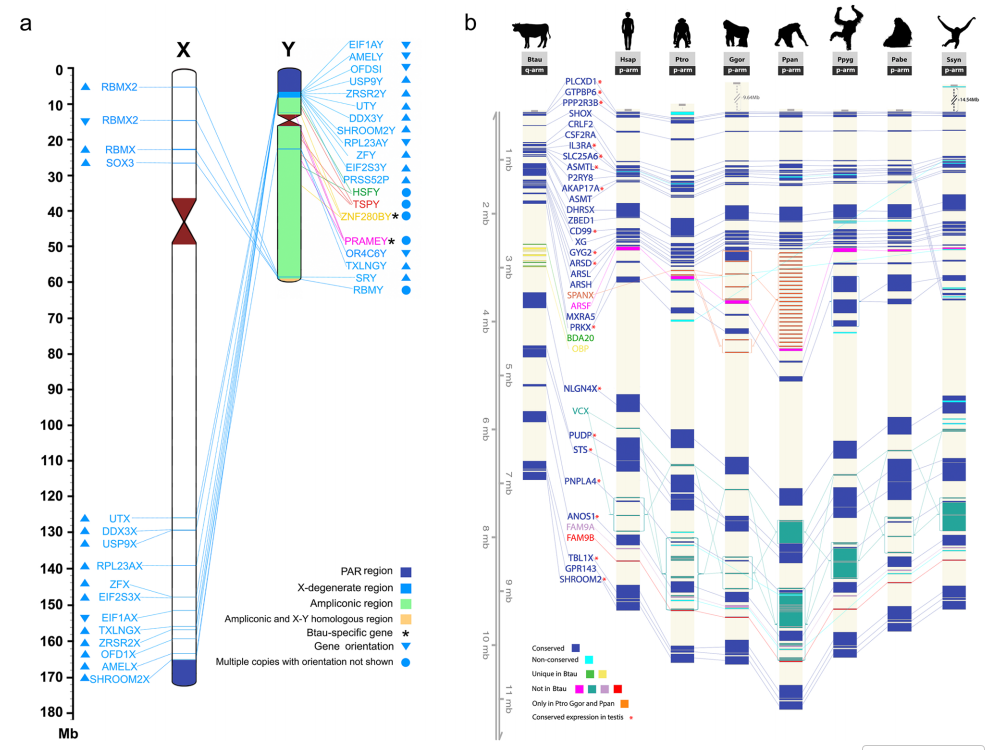
图5:牛X和Y染色体的基因保护
八、保守 PAR 基因与睾丸表达
基因表达方面,牛睾丸中检测到17,000余基因,其中OBP在睾丸表达且幼年更高,而BDA20未在睾丸表达。多种物种中保守的PAR基因(如PPP2R3B、ASMTL、CD99等)在牛中也表现出一致的睾丸表达特征。值得注意的是,TBL1Y虽为所有物种的Y基因,但仅在牛被纳入PAR;牛特有的PRP亦在睾丸表达。
九、发现新的牛基因
作为目前最完整的牛基因组组装,UOA_Wagyu_1_Y的注释结果相比参考基因组显著扩展,共新增10,566个此前未在染色体1-29与X上记录的基因组特征,其中95%为lncRNA、rRNA与蛋白编码基因。X染色体新增的蛋白编码基因数量最多,共337个;其中10个来自PAR区,说明ARS-UCD2.0在该区域存在错误组装。在BTAX着丝粒区域,研究共鉴定37个蛋白编码基因,其中24个为首次发现。GO与通路富集分析显示,这些基因显著富集于“胰岛素调节血糖”等代谢相关通路,提示其可能具有未被认识的调控功能。
十、构建并表征和牛结构变异(SV)目录
本研究采用最大捕获遗传多样性(MCG)方法优先选择最具代表性的种公牛,并逐步加入能补充未覆盖变异的个体,以在既定预算内最大化捕获遗传多样性。结果显示,在选取20头重测序样本时,MCG方法捕获的遗传变异明显多于随机抽样。利用四种结构变异(SV)检测工具对这20个样本进行分析,并分别以UOA_Wagyu_1_Y与ARS-UCD2.0为参考组装,共鉴定到约4.7-5.0万个SV,其中插入与缺失占主要部分,其余依次为倒位、重复和断点;多数SV分布在基因间区,其次为内含子和外显子区域。SV长度以短变异为主,并在约145bp与285bp(SINE-BOV-A2)、1300bp(LTR)及8500bp(LINE/L1)处出现峰值;UOA_Wagyu_1_Y中还存在约2700bp的LINE缺失峰。为识别和牛特异的基因组区域,本研究构建包含13个肉牛品种的泛基因组图,共鉴定到283,348个PAV,其中5840个为和牛特有。整合SV与PAV后共识别133个和牛特异性热点,并发现509个与其重叠的蛋白编码基因,多与嗅觉功能相关。
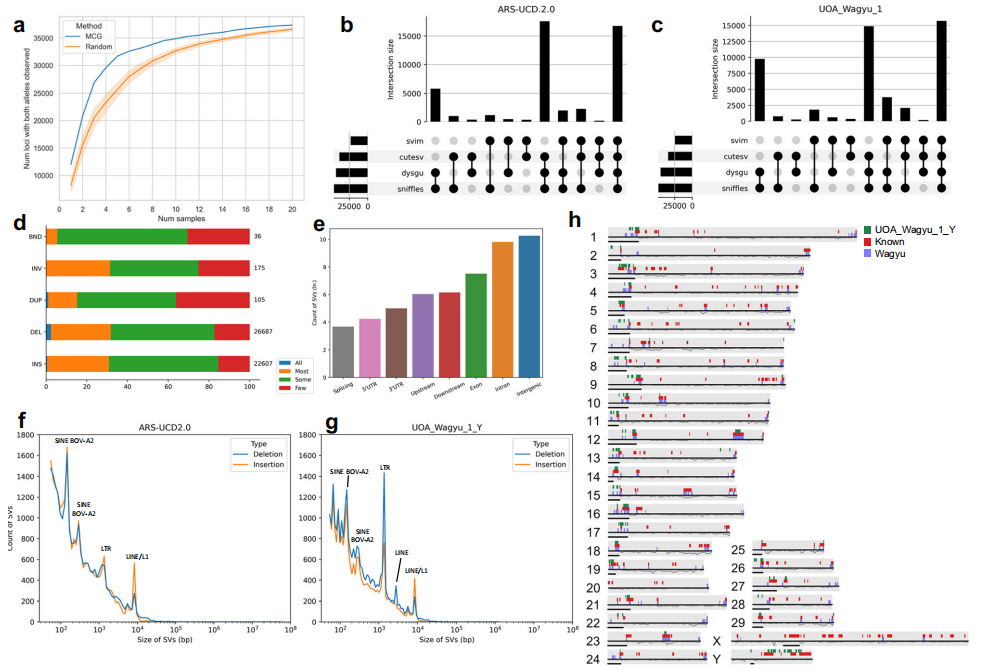
图6:和牛结构变异(SV)目
结语
作者利用PacBio HiFi与ONT超长读长组装出近乎端粒到端粒的和牛基因组UOA_Wagyu_1,完整解析X染色体及4条常染色体,使基因组长度较现参提高16%,大幅补全着丝粒、端粒与rDNA等高度重复区域,并新增大量基因注释(尤其集中在X染色体)。研究解析常染色体着丝粒的7类卫星重复结构,发现牛X染色体着丝粒不同于典型哺乳动物结构,几乎不含卫星DNA,而由倒位重复与转座元件构成,且CENP-A富集、CpG密度与甲基化水平均异常低,揭示其为由转座扩增和CpG脱氨等机制驱动形成的天然新生着丝粒。作者进一步比较多物种X染色体与牛X-Y伪常染色体区,确认多基因在睾丸中具有保守表达模式,并构建20头和牛的结构变异图谱,为性染色体演化研究与育种应用提供高质量参考。

