Trends Plant Sci精选 | 从T2T基因组到超级泛基因组
随着测序成本持续下降及计算方法的快速发展,植物基因组学正迈入新的范式转变。单倍型分相的端粒到端粒(T2T)组装与超级泛基因组的结合,正在成为植物研究所需的“终极参考框架”。二者协同可为遗传解析、分子设计育种以及种质资源保护提供黄金标准。
海南大学陈飞教授于2025年12月1日在期刊Trends in Plant Science上发表标题为“Phased telomere-to-telomere super-pangenome: definitive reference genome in plants”的观点文章,系统性整合并概述了单倍型 T2T 与超级泛基因组在植物基因组学未来发展中的关键驱动力与应用前景。

一、从端粒到端粒(T2T)到单倍型 T2T
传统组装在着丝粒、端粒等高度重复区域仍存在大量缺口,而随着长读长测序与 Hi-C 框架的成熟,染色体得以实现真正意义上的T2T拼接。而单倍型 T2T 更进一步,在实现无缺口组装的同时,能够清晰分离不同亲本来源的单倍型,为结构解析带来前所未有的精细度。最新的 Mo17、甜瓜 821 和草莓 Hawaii 4 基因组体现了这一进展:缺口数量大幅减少,端粒与着丝粒结构完整呈现,TAG重复序列、核仁组织区以及大量先前难以解析的重复序列得以准确恢复。碱基准确度和 BUSCO完整度显著提升,多个物种中新增了大量基因模型,使过去的“基因组黑暗区域”逐渐被照亮。T2T 与单倍型 T2T 的结合,为深入理解植物基因组结构、解析关键变异并支撑精确育种奠定了新的基准。
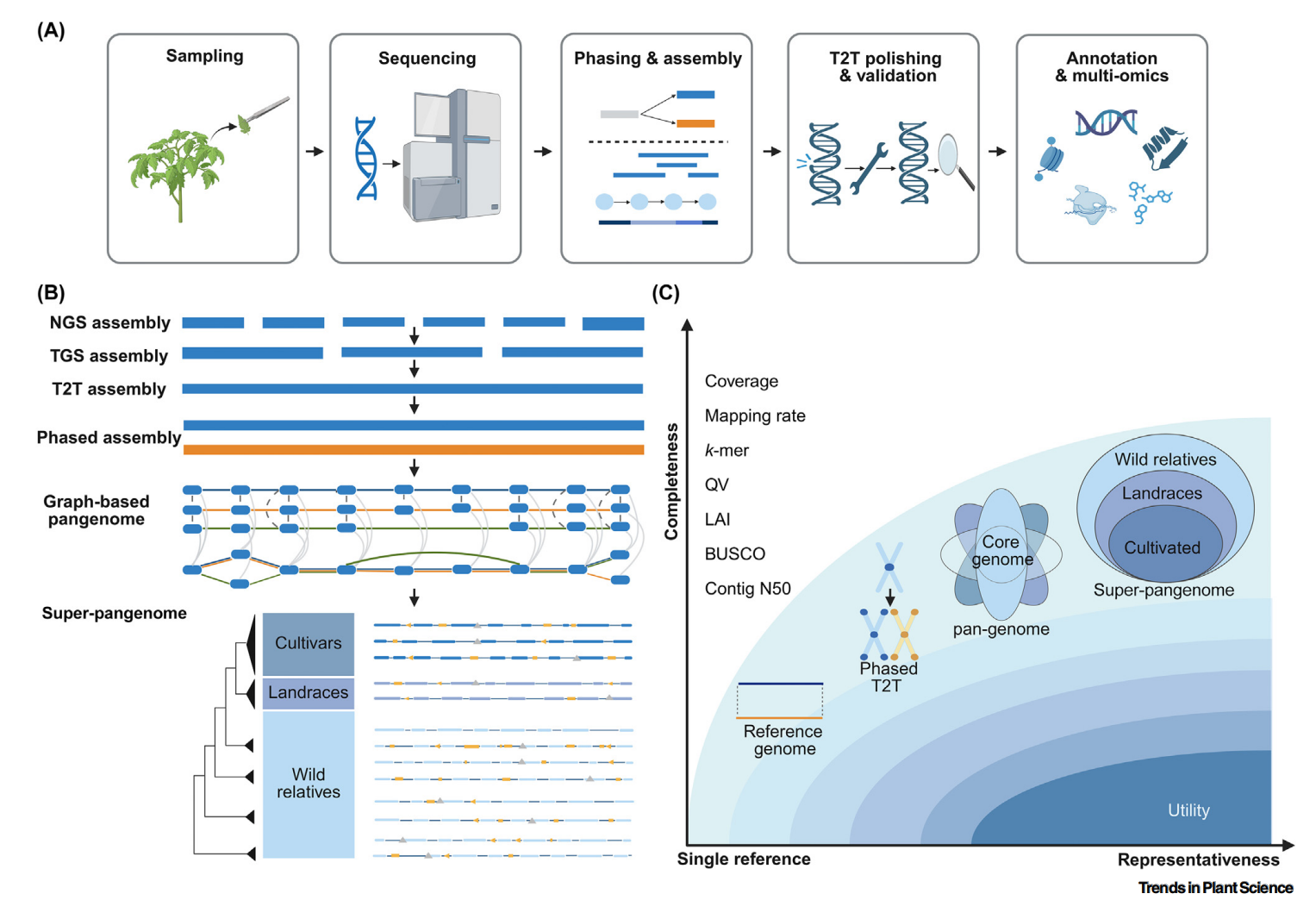
图1:端粒到端粒组装的工作流程和基准
二、从泛基因组到超级泛基因组
泛基因组通过单一参考捕捉种群多样性,但受限于样本量,难以全面反映物种间的遗传变异。最近的研究提出了超级泛基因组的概念,通过整合多个物种的基因组,揭示了更多遗传变异。以番茄为例,13个染色体级基因组构建的超级泛基因组显示,54%的基因在物种间保守,38.4%的基因变异,7.6%的基因特异于某个物种,说明跨物种比较揭示了丰富的遗传变异。在玉米、大豆、水稻等作物中,超级泛基因组的应用推动了基因组研究的进展。例如,大豆的超级泛基因组挖掘出了64124个多年生特有基因,水稻中结合T2T参考的超级泛基因组验证了一个影响分蘖数的着丝粒基因。番茄的研究表明,超级泛基因组揭示了传统GWAS难以捕捉的结构变异和与性状相关的基因,如细胞色素P450基因的缺失与香气和产量的关联。这些研究展示了超级泛基因组在遗传变异解析、功能基因发现和作物改良中的巨大潜力。
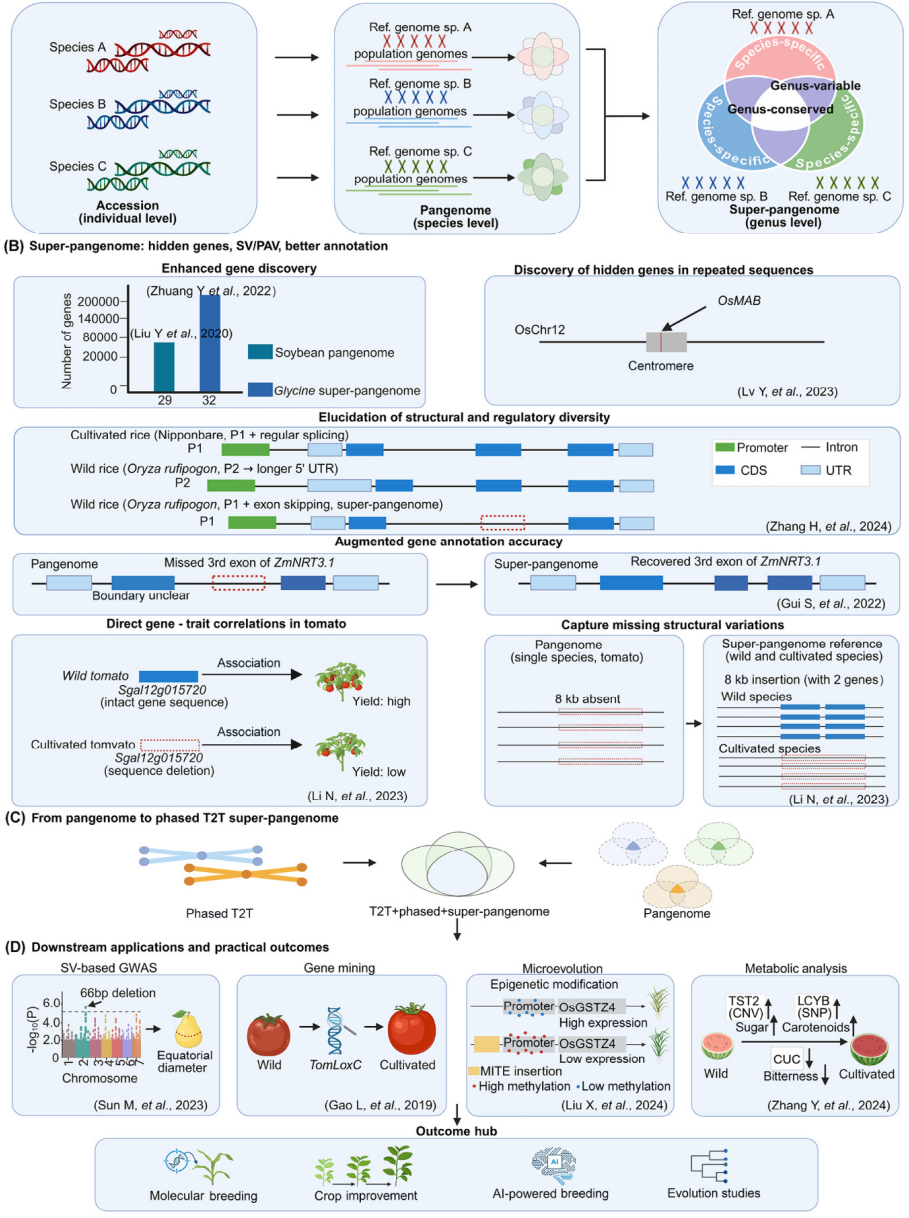
图2:超级泛基因组在隐匿基因发现、结构变异(SV)捕获与育种转化中的应用
三、单倍型T2T与超级泛基因组:终极参考的新时代
单倍型T2T和超级泛基因组的结合为植物基因组学提供了一个全新的参考框架。单倍型T2T确保了单个个体的完整基因组,而超级泛基因组则通过整合多个物种的基因组,揭示了物种级别的遗传多样性,通过这一技术,研究人员能够精确追踪农艺性状的进化轨迹,尤其在多倍体物种中解决了基因组组装和变异检测的挑战。未来,结合第三代长读长测序、单细胞测序和图谱建模的策略,将进一步推动基因组解析和功能基因发现。同时, AI技术的应用也将提高组装精度、可扩展性,并支持超级泛基因组框架的自动化构建和功能注释。随着技术的不断进步,单倍型T2T与超级泛基因组的整合将为作物改良、复杂性状解析和基因功能挖掘提供新的可能。
四、大型T2T与超级泛基因组的构建难点与策略
当前植物基因组研究正迈向 T2T组装与超级泛基因组的深度整合,尽管 PacBio HiFi 与 ONT 超长读长技术不断成熟,实现真正无缺口的基因组组装仍需极高测序深度、强大的内存资源及多层级 Hi-C 或光学图谱,从而带来显著的经济和技术压力。
为兼顾成本与实用性,近年来提出了多种优化策略,如将高保真读长与参考引导组装、部分 Hi-C或光学数据结合,其中 RagTag 等工具已在多物种中成功构建染色体水平组装,这类混合与图结构方法无需完全投入 T2T 仍能显著提升基因组连续性。从应用角度看,组装策略的选择取决于研究目标:若用于功能基因挖掘或性状相关结构变异检测,高完整度组装与超级泛基因组图已能满足需求;而在研究多倍体进化、亚基因组剂量或染色体重排等需高分辨率单倍型解析的场景中,单倍型 T2T 则不可替代。进一步地将单倍型 T2T 参考与跨物种超级泛基因组整合,不仅能增强变异解释能力,还可提升基于结构变异的 GWAS、等位基因特异性表达分析及代谢适应研究的效率。
五、对育种与资源保护的意义
随着植物基因组学的发展,单倍型 T2T 组装与超级泛基因组的结合已成为推动育种和生物多样性保护的关键。单倍型 T2T 组装能够精确解析基因剂量效应和等位基因调控,为分子设计育种提供高分辨率的基因组数据;而超级泛基因组则整合了来自野生近缘种的功能序列,能够在引入优势性状的同时最小化不良连锁基因的风险。例如,野生番茄中细胞色素 P450 基因的过表达可使栽培种产量提高 67.1%。在物种保护方面,随着气候变化和极端环境的挑战日益严峻,超级泛基因组融合野生多样性,为基因组编辑、基因组选择和抗逆新品种的培育提供了精准靶点。随着技术进步,植物基因组学正从单一参考基因组迈向“单倍型 T2T+超级泛基因组”的新阶段,这一趋势将重塑未来植物遗传、育种与物种保护研究的基础。
结语
本文探讨了将单倍型 T2T 基因组与超级泛基因组相结合所引领的植物基因组学新趋势。单倍型T2T级别的组装显著提升了基因组解析的精度与完整性;而超级泛基因组则通过整合多物种或多群体的遗传多样性,揭示了传统参考基因组中缺失的隐匿基因和结构变异,为功能基因挖掘与复杂性状解析提供了更具深度的遗传框架。将 T2T 基因组与超级泛基因组相结合,可构建真正“无缺口、无盲区、无偏差”的基因组参考体系,大幅提升精准育种和基因编辑的可操作性与效率。

